一杯DNA裝下全世界?MIT突破DNA新檢索技術,實現DNA「冷存儲」
一個咖啡杯就能裝下全世界?
有了DNA數據存儲,這是可能的。
1988年,藝術家Joe Davis和哈佛大學研究人員合作,首次證明了DNA存儲數字化數據的原理。
Davis通過明暗像素將代表35bits數據的符文符號圖像表示為二進制0和1,并將其編碼成了大腸桿菌DNA中的28個堿基對。
隨后,存儲在DNA中的數據也從簡單文本變成高清音樂視頻、整個數據庫、MPEG、JPG、PDF等文件,甚至還有惡意軟件。
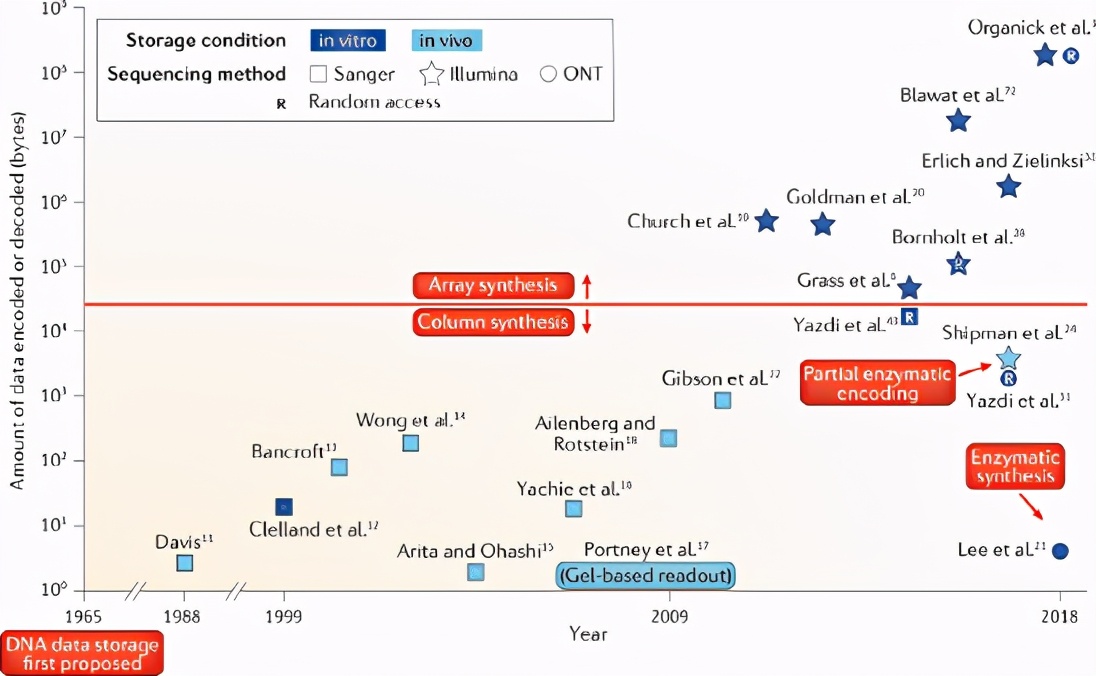
DNA數據存儲發展過程(1965-2018)(圖源:nature)
DNA數據存儲是什么
DNA數據存儲是一個將二進制數據轉換成人工合成DNA鏈的編碼過程。
為了在DNA中存儲二進制數字文件,比特(bits)將從1和0轉換成字母A,C,G,T,這四個字母代表組成DNA的四種核苷酸:腺嘌呤,胞嘧啶,鳥嘌呤,胸腺嘧啶。
物理存儲介質是一條序列中包含As, Cs, Gs, Ts的合成DNA鏈,其順序與數字文件中的bits相對應,如果要恢復數據,需要對DNA鏈進行測序,根據As, Cs, Gs, Ts還原成初始的數字序列。
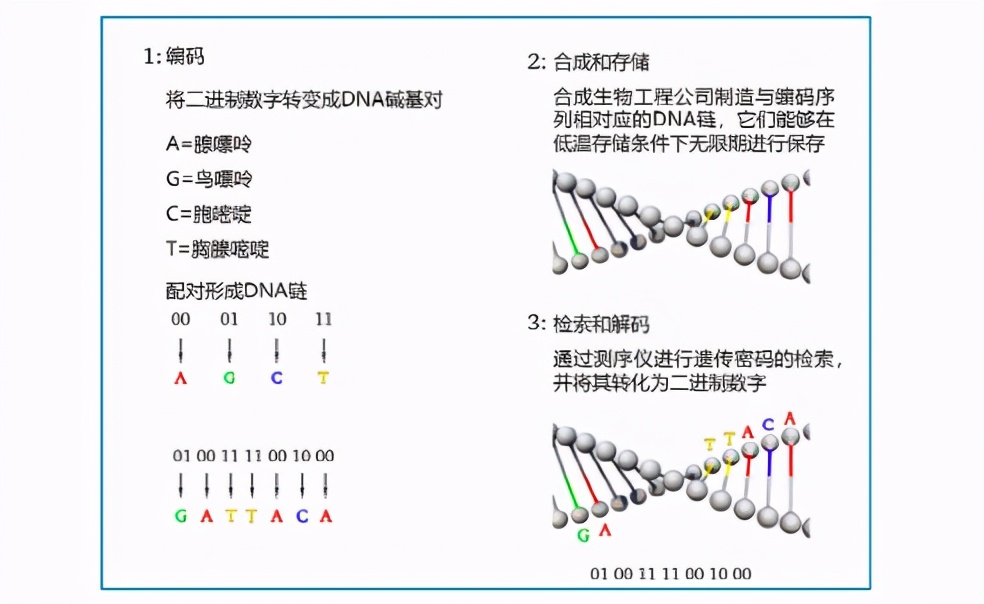
數字化的數據在DNA中編碼和解碼的過程(圖源:https://www.ssbt.org.cn/upload/20190905163302_429.pdf)
在人類創造數據能力不斷增長的今天,基于DNA的數據存儲似乎是個「潛力股」。
因為與其他大多數媒介相比,DNA提供了驚人的「數據存儲密度」,而且相比傳統數據存儲,它具有高度穩定性:DNA分子半衰期超過「500年」,低溫條件下可保存「成千上萬年」。
但DNA并非沒有缺點,成本高昂是阻礙其發展的主要問題。
目前,DNA鏈的堿基模式中沒有編碼比特的標準方法,合成特定的序列仍然很昂貴。而用目前的方法訪問數據不僅慢,而且會消耗用于存儲的DNA。如果試圖訪問數據的次數太多,就必須以某種方式恢復它,這有可能引入錯誤。
近日,麻省理工學院和Broad研究所(Broad Institute)的一個團隊找到了一個解決方案。在這個過程中,研究人員創建了一個基于DNA的圖像存儲系統,它介于「文件系統」和「基于元數據的數據庫」之間,相關論文已在Nature上發表。

把所有數據存儲到DNA上的瓶頸
在DNA中存儲數據的系統涉及到向包含數據的DNA片段添加特定的序列標簽。
為了得到想要的數據,你只需添加能與正確的標簽堿基配對的DNA位,并使用它們來擴增完整的序列。可以把它想象成用一個 ID 標記集合中的每個圖像,然后進行設置,只放大一個特定的 ID。

這種方法是有效的,但它有兩個方面的限制。
首先,使用稱為PCR(聚合酶鏈式反應)的過程進行的擴增步驟,對可擴增的序列的大小有限制。而每個標簽都會占用一些有限的空間,所以添加更多詳細的標簽(如復雜的文件系統可能需要)會減少數據空間。
一條 8 個 PCR 管,每個管含有 100 μL 反應混合物
另一個限制是,擴增特定數據片段的 PCR 反應會消耗一些原始的 DNA 庫。換句話說,每次你拉出一些數據,你都會破壞成堆的不相關的數據。頻繁地訪問數據,最終會耗盡整個存儲庫。雖然有辦法重新放大一切信息,但每次這樣做都會增加引入錯誤的機會。
而這項新的研究已經將標簽信息從數據存儲中分離出來。此外,研究人員創建了一個系統,其中可以只訪問你感興趣的DNA數據,而不觸及其余的數據,提高了數據存儲的壽命。
給二氧化硅磁珠添加「涂層」
該基本技術是基于這樣一個事實,即DNA會粘在二氧化硅磁珠(beads)上。
但這種吸力與DNA的大小無關,因此你可以使用這個系統存儲任意大的數據塊(在這種情況下,這些片段的大小是過去使用的典型的DNA數據存儲塊的10倍以上)。
同樣重要的是,DNA中沒有標簽被存儲在數據中,所以數據存儲和文件系統信息之間沒有競爭。
一旦DNA出現在這些磁珠的表面,研究人員就在其上面聚合一些額外的二氧化硅。這個過程涂抹了DNA并保護它不受環境影響。

研究人員通過使用熒光標簽來確認該系統是有效的;基本上,所有以這種方式創造的顆粒都含有DNA。

只有當這個外殼就位后,研究人員才添加標簽,這些標簽與外殼進行化學連接。這些標簽是由單鏈DNA制成的,而且有可能在一個玻璃外殼上附著幾個不同的標簽。
研究人員對每個數據塊分別進行了處理,一旦一切就緒,被標記的玻璃球就可以混入一個單一的數據庫。
雖然沒有純DNA的存儲那么緊湊,但仍然具有長期穩定和不需要能源維護的優勢。
取代PCR
有趣的部分是訪問數據。
除了成本之外,使用DNA存儲數據的另一個主要瓶頸是,很難從所有文件中挑選出想要的文件。
此次開發的新的檢索技術,希望取代PCR方法。
研究人員將每個DNA文件封裝到一個微小的二氧化硅磁珠中,每個磁珠都貼上了由單鏈DNA組成的「條形碼」,與文件內容相對應。
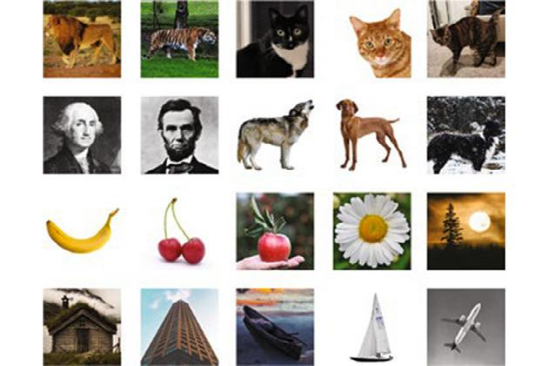
為了證明這種方法的成本效益,研究人員將20個不同的圖像編碼到大約長度為3000個核苷酸的DNA片段中,這大致相當于100個字節(研究還顯示,這些磁珠可以容納高達1GB的DNA文件)。
研究中的每個文件都有相應的條形碼標簽,如「貓」或「飛機」等。
當研究人員想要提取一個特定的圖像時,他們會取出一個DNA樣本,加入與目標標簽相對應的引物。例如,老虎的圖像對應的標簽是「貓」「橘色」和「野生」,而家貓的圖像對應「貓」「橘色」和「家養」。
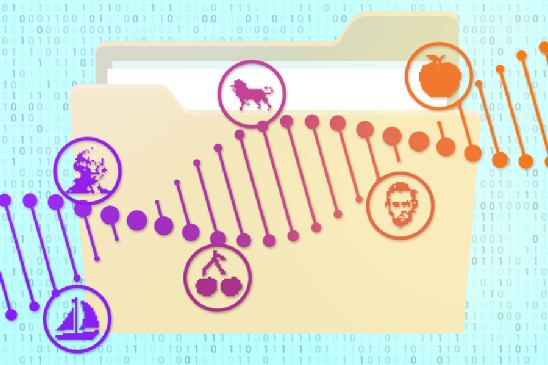
這些引物用熒光或磁性顆粒標記,便于從樣本中提取并識別匹配片段。
通過這種方法,研究人員可以將需要的文件移出來,剩下的DNA則完整地放回去,繼續存儲數據。
他們的檢索過程允許「布爾邏輯語句」,如「總統和18世紀」會生成「喬治·華盛頓」的結果,這很類似谷歌的圖像檢索。
在目前的概念驗證階段,搜索速度是每秒1000字節(1KB)。文件系統的搜索速度是由每個磁珠的數據量大小決定的,而目前限制數據量大小的因素就是在DNA上寫入100兆字節(MB)數據所需的高昂成本,以及可以并行使用的分類器的數量。
如果DNA合成變得足夠便宜,就能夠用這種方法將每個文件存儲的數據量最大化
DNA數據存儲目前局限于「冷存儲」
該系統還允許用多個術語進行「布爾搜索」(Boolean search)。
通過一個接一個地選擇不同的標簽,你可以建立起相當復雜的條件:貓為真,馴養的為假,黑為真,等等。
給兩個標簽貼上相同的熒光顏色,如果你抓到任何帶有這種顏色的東西,你就可以得到相當于邏輯OR的結果。
因為這些標簽中的每一個都可以被看作是關于DNA所存儲的圖像的元數據,磁珠的集合最終作為一個元數據驅動的圖像數據庫。
雖然這項研究代表了基于DNA的存儲在復雜性方面的一個重大飛躍,但它仍然只是基于DNA的存儲。
這意味著它的速度之慢,甚至還不如磁帶驅動器。

根據研究人員的計算,即使他們把更多的數據塞進每顆磁珠,搜索上限只是每秒約1GB的數據。這將意味著搜索PB級的數據將需要「兩周多」的時間。
而這僅僅是找到合適的磁珠。敲開它們,將DNA放進去,然后進行必要的測序,以實際確定磁珠中儲存的內容,這又會使實驗過程增加幾天。
當然,沒有人會因為DNA存儲「速度快」而推薦它;正如上面提到的,它的優勢在能源使用和數據穩定性方面。
我們只有在確定不會經常訪問某些數據時才會將它儲存在DNA中,也即「冷存檔存儲」。
不過,目前,該實驗室已經成立了一家名為Cache DNA的初創公司,正在開發DNA的長期存儲技術,既可以用于長期的DNA數據存儲,也能用于短期的臨床和其他現有的DNA樣品存儲。

https://www.cache-dna.com/
雖然可能還需要一段時間才能將DNA作為數據存儲介質,但目前在Covid-19檢測、人類基因組測序和其他基因組學領域中,對于DNA和RNA樣品的低成本和大規模存儲的解決方案都有很大需求。






























