頂尖科學家如何玩轉AI?DeepSpeed4Science:利用先進的AI系統優化技術實現科學發現
在接下來的十年中,深度學習可能會徹底改變自然科學,增強我們對自然現象進行建模和預測的能力。
這可能預示著科學探索的新時代,為從藥物開發到可再生能源的各個領域帶來重大進展。
對此,微軟DeepSpeed團隊啟動了一個名為DeepSpeed4Science的新計劃,旨在通過AI系統技術創新幫助領域專家解鎖當今最大的科學之謎。
DeepSpeed系統是由微軟開發的業界領先的開源AI系統框架,它為各種AI硬件上的深度學習訓練和推理提供了前所未有的規模和速度。
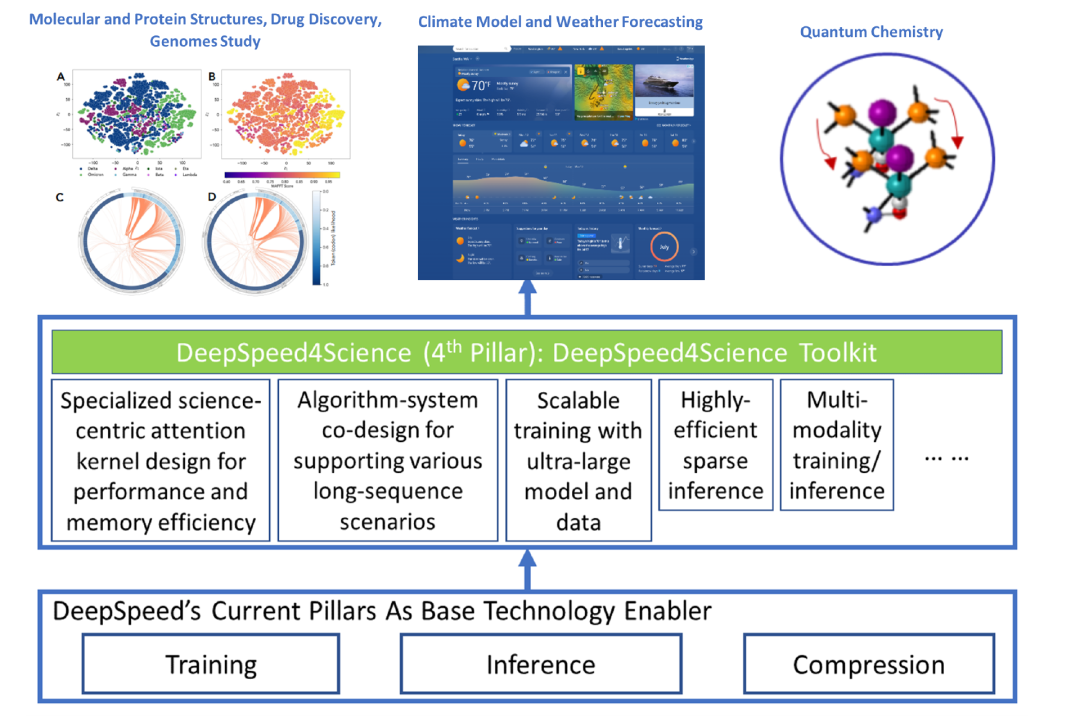
圖1:DeepSpeed4Science方法概述:專為加速科學發現和應對其復雜性而量身定制的AI系統技術開發。
圖1展示了我們對DeepSpeed4Science這一新計劃的基本方法。
通過利用DeepSpeed當前的技術方案(訓練、推理和壓縮)作為基礎技術推動器,DeepSpeed4Science將創建一套專為加速科學發現而量身定制的AI系統技術,以應對其獨特的復雜性,超越用于加速通用大型語言模型(LLMs)的常見技術方法。
在這篇博客中,我們展示了DeepSpeed4Science如何幫助解決結構生物學研究中的兩個關鍵AI系統挑戰:
(1)解決了以Evoformer為中心的蛋白質結構預測模型中的內存爆炸問題,以及
(2)為更好地理解引發大流行的疾病的進化提供AI模型長序列支持。
我們的初期主要合作者
DeepSpeed4Science的新系統技術可以用于很多推動科學邊界的標志性模型,賦能AI驅動的科學發現。
目前,DeepSpeed4Science很榮幸地支持來自微軟研究院AI4Science、微軟WebXT/Bing、美國能源部國家實驗室和多所大學的幾個關鍵科學模型。
內部合作伙伴
科學基礎模型(Scientific Foundation Model,SFM),微軟研究院AI4Science
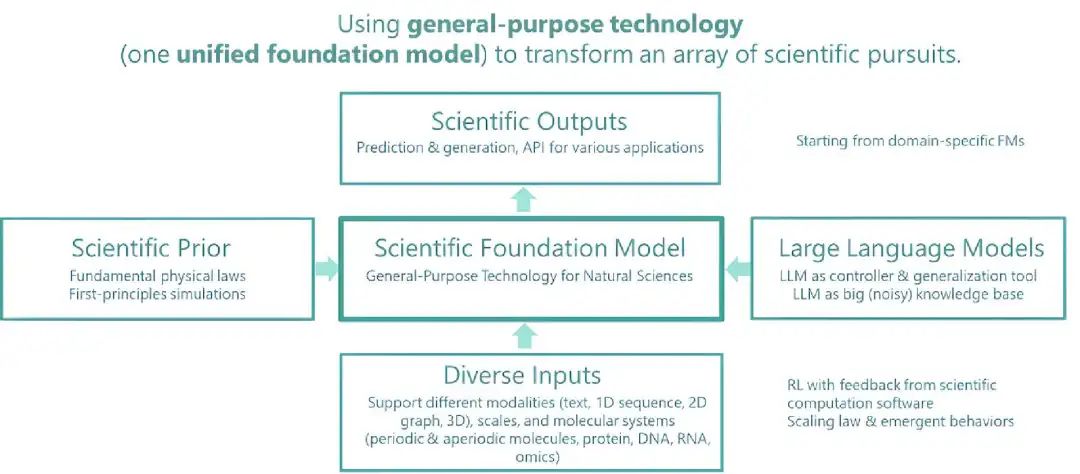
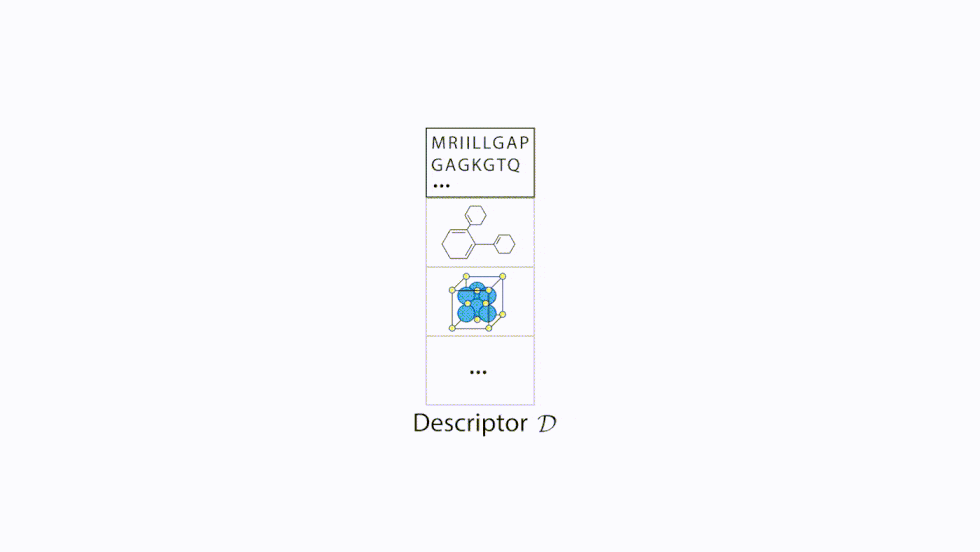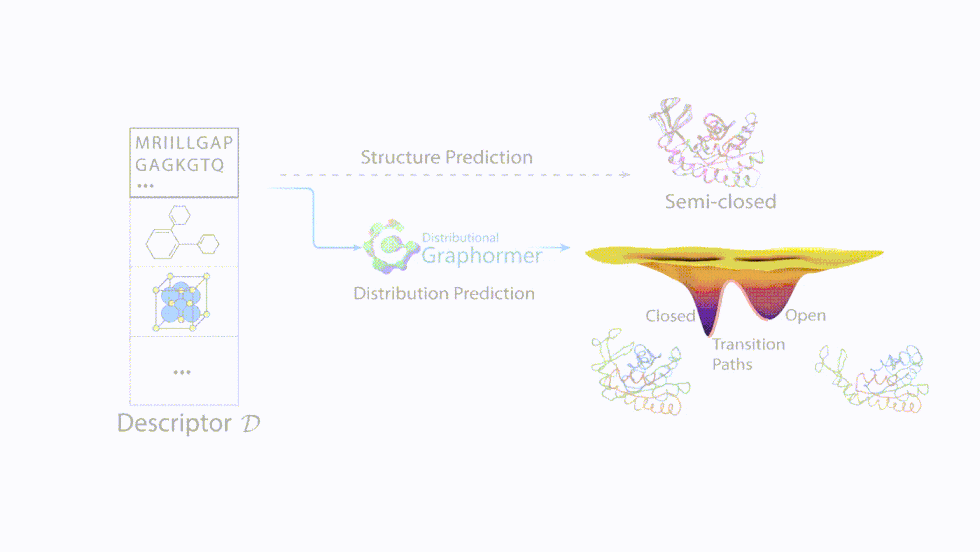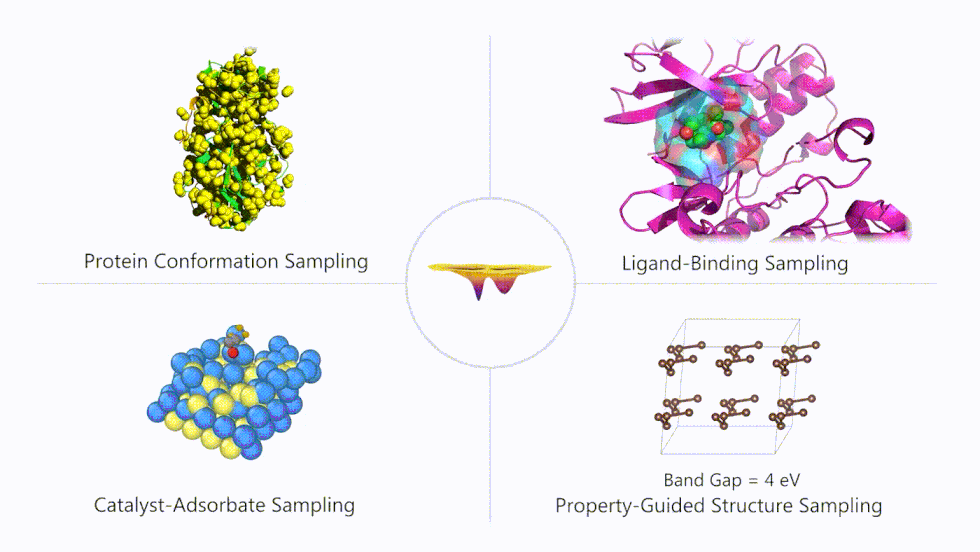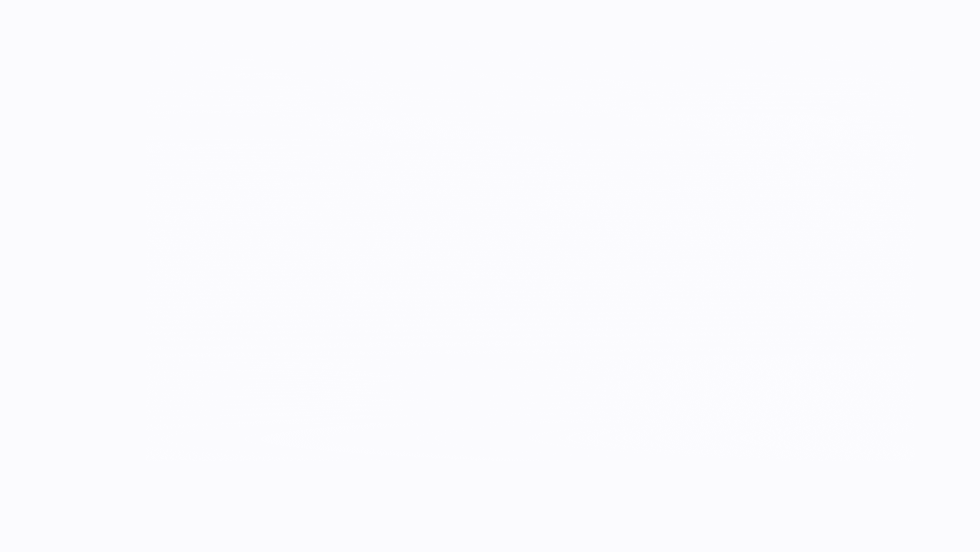
圖2:科學基礎模型(Scientific Foundation Model,SFM)及其當前探索:Distributional Graphormer
科學基礎模型(SFM)旨在創建一個統一的大規模基礎模型,以支持自然科學發現,支持多種輸入、多個科學領域(例如,藥物、材料、生物學、健康等)和計算任務。
DeepSpeed4Science合作伙伴關系將為SFM團隊提供新的訓練和推理技術,以支持他們的新生成AI方法(例如Distributional Graphormer)這樣的項目進行持續研究。
ClimaX,微軟研究院AI4Science
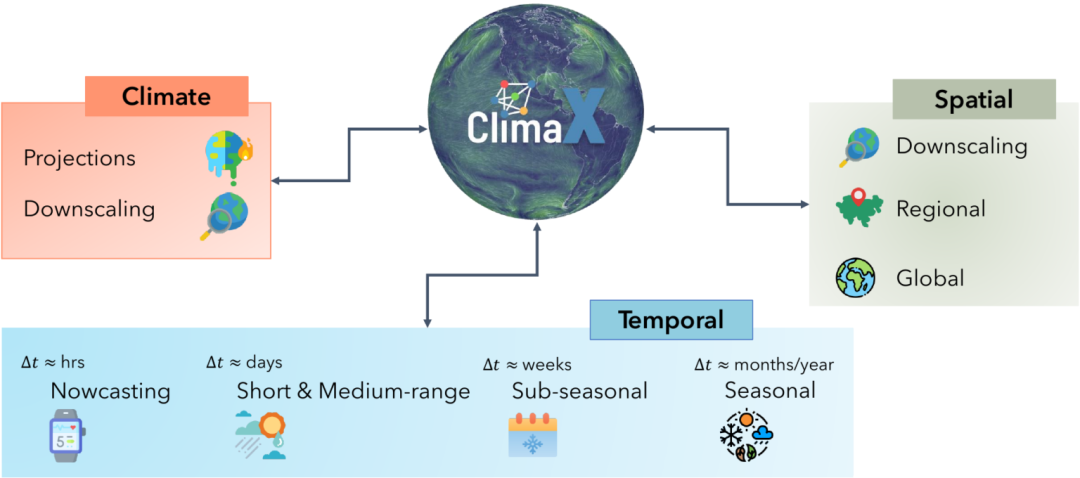
圖3:ClimaX是第一個設計用于執行各種天氣和氣候建模任務的基礎模型
我們的氣候正在發生變化,導致極端天氣事件的頻率增加。為了減輕負面影響,預測這些事件將發生的地方變得越來越重要。
ClimaX是第一個設計用于執行各種天氣和氣候建模任務的基礎模型。它可以吸收許多具有不同變量和分辨率的數據集以提高天氣預報的準確性。
DeepSpeed4Science正在為ClimaX創建新的系統支持和加速策略,以高效地預訓練/微調更大的基礎模型,同時處理非常大的高分辨率圖像數據(例如,數十到數百PB)和長序列。
AI驅動的第一性原理分子動力學(AI Powered Ab Initio Molecular Dynamics,AI2MD),微軟研究院AI4Science
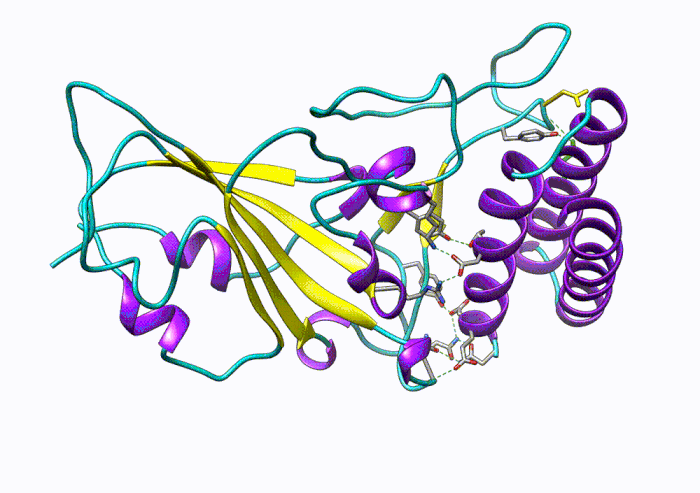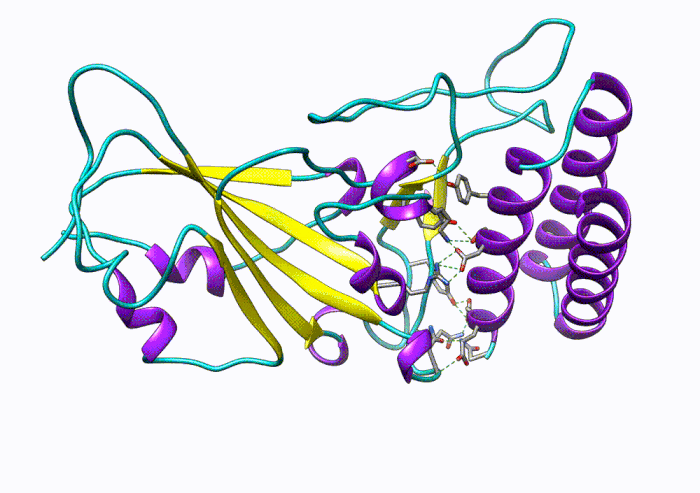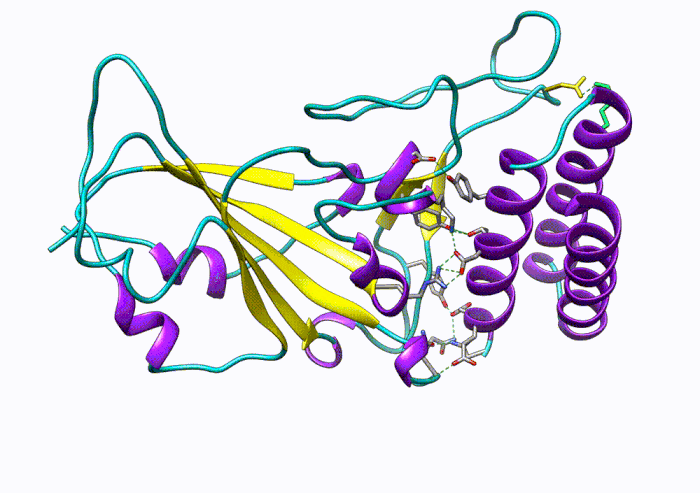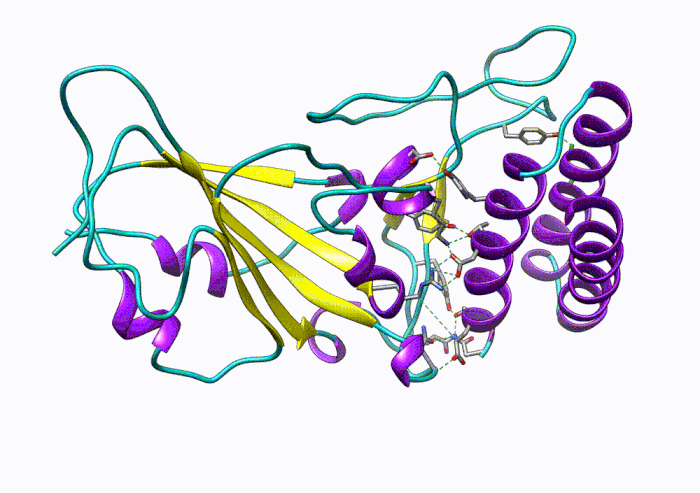
圖4:一百萬步的分子動力學模擬:RBD-蛋白(RBD-protein)與蛋白抑制劑(protein inhibitor)相互作用。
這個項目模擬了使用AI驅動的力場模型進行近似第一性原理計算精度的大型(百萬原子)分子系統的動態模擬,同時保持了經典分子動力學的效率和可擴展性。這些模擬足夠高效,可以生成足夠長的軌跡來觀察化學上有意義的事件。
通常,這個過程需要數百萬甚至數十億的推理步驟。這對優化圖神經網絡(GNN)+ LLM模型的推理速度提出了重大挑戰,DeepSpeed4Science將為此提供新的加速策略。
微軟天氣,微軟WebXT/Bing
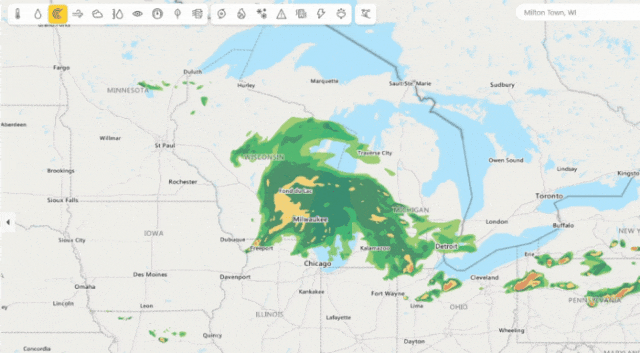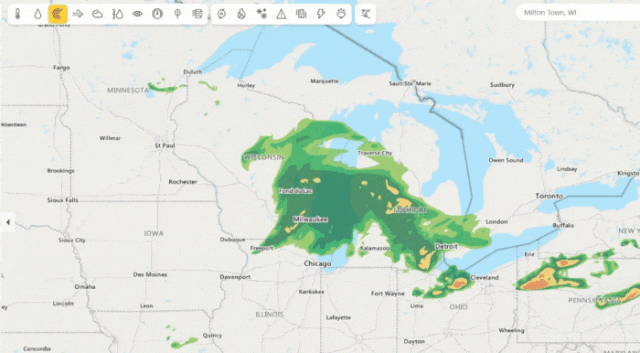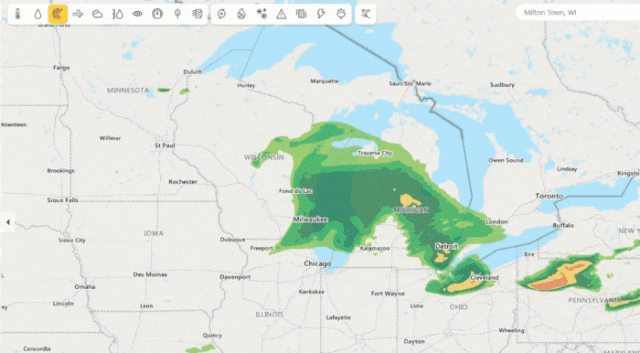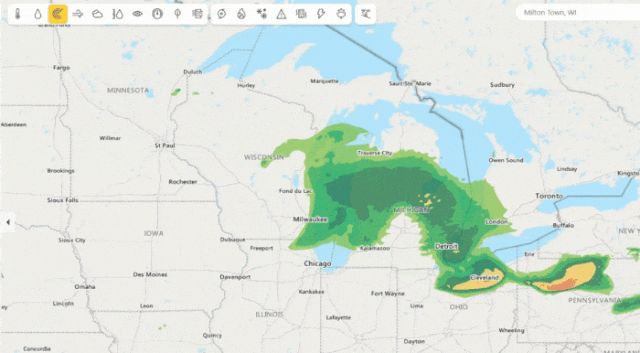
圖5:微軟降水預報(每4分鐘一次對接下來4小時進行預測)。
微軟天氣提供精確的天氣信息,幫助用戶為他們的生活方式、健康、工作和活動做出更好的決策——包括每小時多次更新的準確的10天全球天氣預報。
此前,微軟天氣受益于DeepSpeed技術,加速了他們的多GPU訓練環境。
現在,DeepSpeed4Science正在與微軟WebXT天氣預報團隊合作,進一步增強微軟天氣預報服務的最新功能和改進。
外部合作者
DeepSpeed4Science的旅程始于兩個開創性的基于LLM的結構生物學研究AI模型:來自哥倫比亞大學的OpenFold,一個開源的高保真蛋白質結構預測模型;以及來自阿貢國家實驗室的GenSLMs,一個獲得ACM戈登貝爾獎的用于學習SARS-CoV-2(COVID-19)基因組的進化的語言模型。
作為此次發布的特色展示,它們代表了當今AI驅動的結構生物學研究面臨的兩個常見AI系統挑戰。我們將在下一節中討論DeepSpeed4Science如何賦能這些科學研究。
此外,DeepSpeed4Science最近擴大了其范圍,以支持更多樣的科學模型。
例如,在我們與阿貢國家實驗室合作訓練Aurora Exascale系統上的萬億參數科學模型的工作中,DeepSpeed4Science技術將幫助他們達到這一關鍵任務所需的性能要求和可擴展性。
此外,通過與橡樹嶺國家實驗室和國家癌癥研究所(NCI)合作進行癌癥監測,DeepSpeed4Science將幫助從非結構化的臨床文本中高保真地提取和分類信息,以供MOSSAIC項目使用。
Brookhaven國家實驗室還將采用DeepSpeed4Science技術,支持使用LLMs開發大型數字雙胞胎模型,以便為清潔能源研究產生更真實的模擬數據。您可以在deepspeed4science.ai上找到有關我們外部合作者及其科學任務的更多詳細信息。
合作展示
展示(I):DeepSpeed4Science通過DS4Sci_EvoformerAttention消除以Evoformer為中心的結構生物學模型的內存爆炸問題
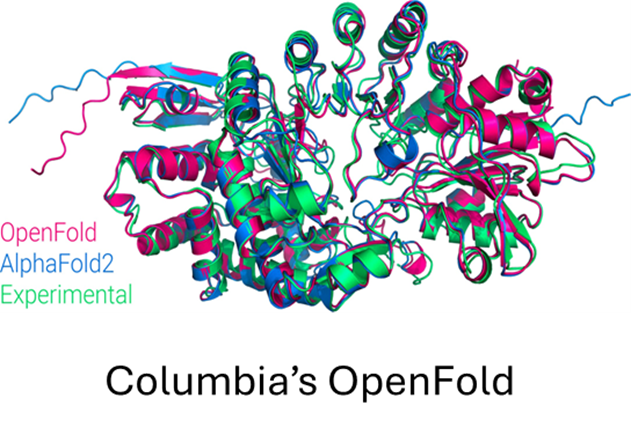

圖6:在訓練過程中OpenFold對PDB鏈7B3A_A的預測
OpenFold是DeepMind的AlphaFold2的開源社區再現,使其可以在新數據集上訓練或微調AlphaFold2。
研究人員已經使用它從頭開始重新訓練AlphaFold2,生成新的模型參數集,研究AlphaFold2的早期訓練階段(圖6),并開發新的蛋白質折疊系統。

圖7:在OpenFold中,對多序列比對(MSA)Attention內核(包含偏差)變體的訓練峰值內存需求。(左)使用在AlphaFold2中的EvoformerAttention的原始OpenFold實現。對于這些類型的蛋白質結構預測模型,在訓練/推理中的內存爆炸問題是常見的。最先進的FlashAttention無法有效支持這樣的Attention變體。(右)DeepSpeed4Science的一種新解決方案DS4Sci_EvoformerAttention在不影響模型品質的條件下顯著地減少了OpenFold的訓練峰值內存需求(最多13倍)。
盡管OpenFold有使用最先進的系統技術進行性能和內存優化,但從頭開始訓練AlphaFold2仍然在計算上很昂貴。目前階段的模型參數很小,只有9300萬個參數,但它包含了幾個需要非常大的中間內存的特殊Attention變體。
在標準AlphaFold2訓練的「微調」階段,只是這些變體中的其中一個在半精度下就生成了超過12GB的張量,使其峰值內存要求遠遠超過了相同大小的語言模型。
即使使用像activation checkpointing和DeepSpeed ZeRO優化這樣的技術,這種內存爆炸問題仍然嚴重限制了可訓練模型的序列長度和MSA深度。
此外,近似策略可能會顯著影響模型的準確性和收斂性,同時仍然導致內存爆炸,如圖7左側(橙色)所示。
為了應對結構生物學研究(例如,蛋白質結構預測和平衡分布預測)中的這一常見系統挑戰,DeepSpeed4Science通過為這類科學模型中廣泛出現的注意力變體(即EvoformerAttention)設計定制的精確注意力內核來解決這一內存效率問題。
具體來說,我們設計了一套由復雜的融合/矩陣分塊策略和動態內存減少方法而組成的高內存效率DS4Sci_EvoformerAttention內核,作為高質量機器學習模塊供更廣泛的生物學研究社區使用。
通過整合到OpenFold中,這些定制內核在訓練期間提供了顯著的加速,并顯著減少了模型的訓練和推理的峰值內存需求。
這使得OpenFold可以用更大、更復雜的模型,使用更長的序列在更廣泛的硬件上進行實驗。關于這項技術的詳細信息可以在這里找到。
展示(II):DeepSpeed4Science通過系統和算法方法為基因組基礎模型(例如,GenSLMs)提供長序列支持
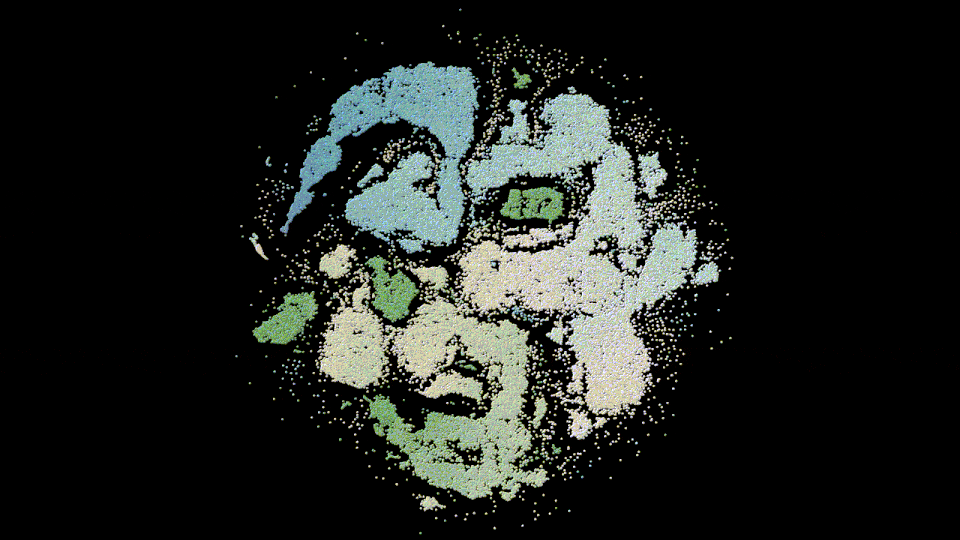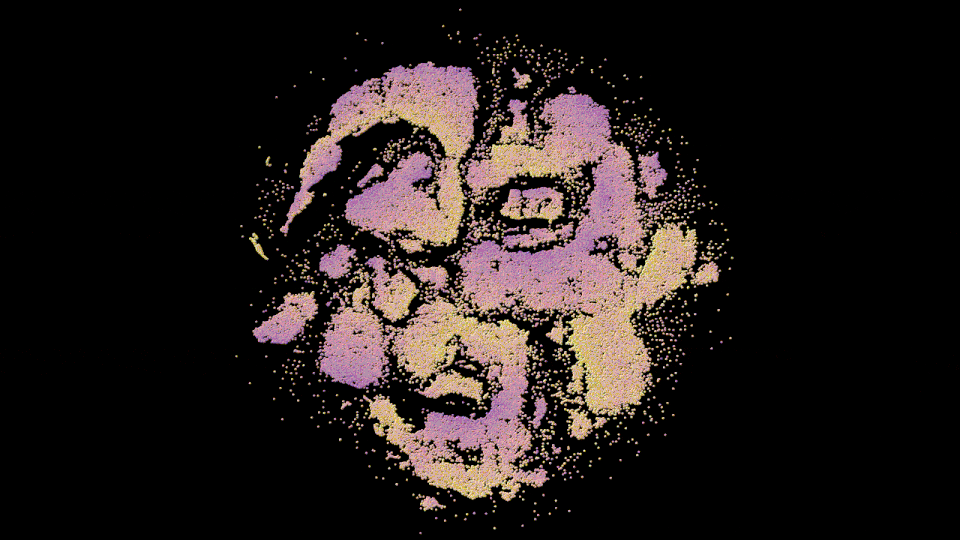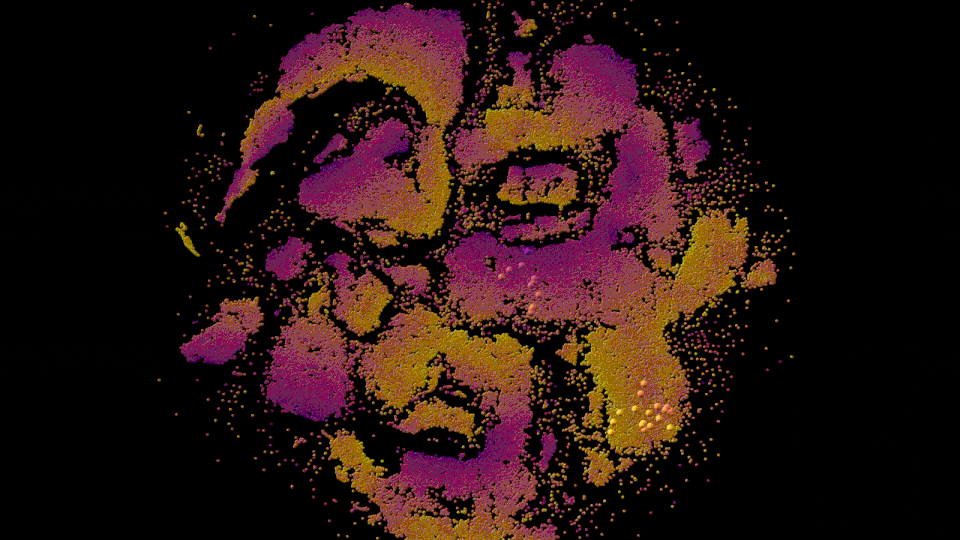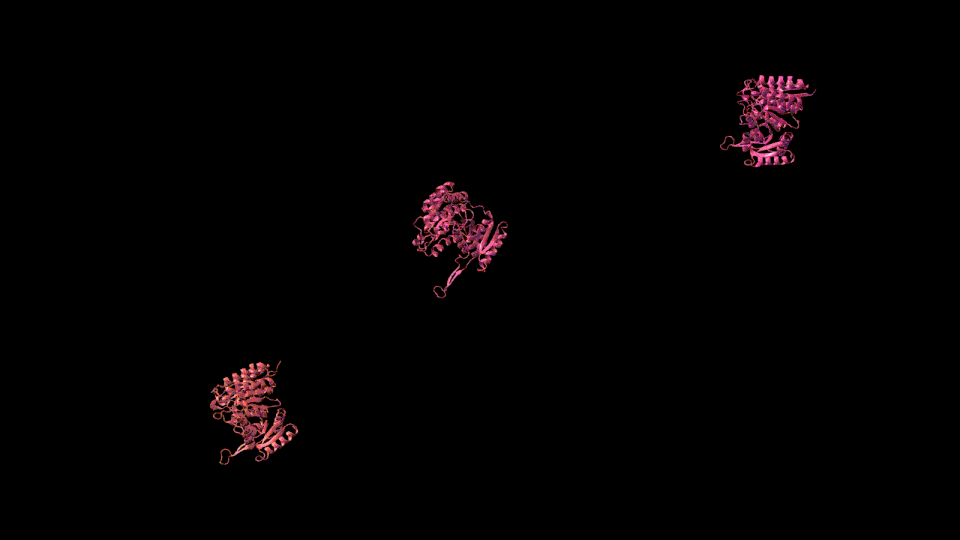
圖8:GenSLMs:獲2022年ACM 戈登貝爾獎的COVID基因組模型(基于GPT-NeoX的25B/33B模型)。它用于學習描述SARS-CoV-2基因組生物學意義的潛在空間。這個GIF展示了一個重要的蛋白質家族蘋果酸脫氫酶(malate dehydrogenase)的根據重要特征(如序列長度和GC含量(核酸鳥嘌呤和胞嘧啶的含量與腺嘌呤和胸腺嘧啶的比率。它測量DNA鏈抵抗熱的能力))著色的潛在空間的投影。
GenSLMs,一個來自阿貢國家實驗室的2022年ACM 戈登貝爾獎獲獎的基因組模型,可以通過大型語言模型(LLMs)的基因組數據訓練來學習SARS-CoV-2(COVID-19)基因組的進化。它旨在改變如何識別和分類引發大流行的病毒(特別是SARS-CoV-2)的新變種。
GenSLMs代表了第一批可以泛化到其他預測任務的基因組基礎模型。對潛在空間的良好理解可以幫助GenSLMs處理超出僅僅是病毒序列的新領域,并擴展它們模擬細菌病原體甚至真核生物的能力(例如,理解功能、途徑成員資格和進化關系等事物)。
為了實現這一科學目標,GenSLMs和類似的模型需要非常長的序列支持用于訓練和推理,這超出了像FlashAttention這樣的通用LLM的長序列策略。
通過DeepSpeed4Science的新設計,科學家現在可以構建和訓練具有顯著更長的上下文窗口的模型,允許他們探索以前無法訪問的關系。
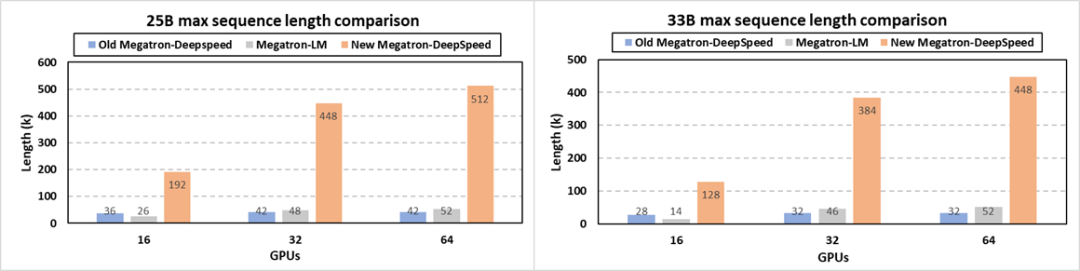
圖9:由不同框架在不同規模下支持的兩個GenSLMs模型的最大序列長度。使用NVIDIA DGX,每個節點有八個40G A100 GPU
具體在系統層面,我們發布了包括長序列支持和其他新優化的最新的Megatron-DeepSpeed框架。
科學家現在可以通過我們新添加的內存優化技術(如注意力掩碼異步處理和位置碼分割)、張量并行、流水線并行、序列并行、基于ZeRO的數據并行和模型狀態異步處理等技術的協同組合,用更長的序列訓練他們的GenSLMs等大型科學模型。
圖9展示了我們的新版本使GenSLMs的25B和33B模型的最長序列長度分別比之前的Megatron-DeepSpeed版本增加了12倍和14倍。
在支持的序列長度方面,這個新Megatron-DeepSpeed框架也顯著地超過了NVIDIA的Megatron-LM(對于25B和33B模型分別高達9.8倍和9.1倍)。
例如,阿貢實驗室團隊的GenSLMs 25B模型在64個GPU上的原始序列長度為42K,而現在可以用512K的核苷酸序列進行訓練。這在不損失準確性的條件下大大提高了模型質量和科學發現的范圍。
對于那些更喜歡相對位置編碼技術這樣的算法策略的領域科學家,這個新版本也進行了集成。
轉載自微軟DeepSpeed組官方知乎賬號:
zhihu.com/people/deepspeed