CVPR 2024 Oral:生命之樹大模型 原創
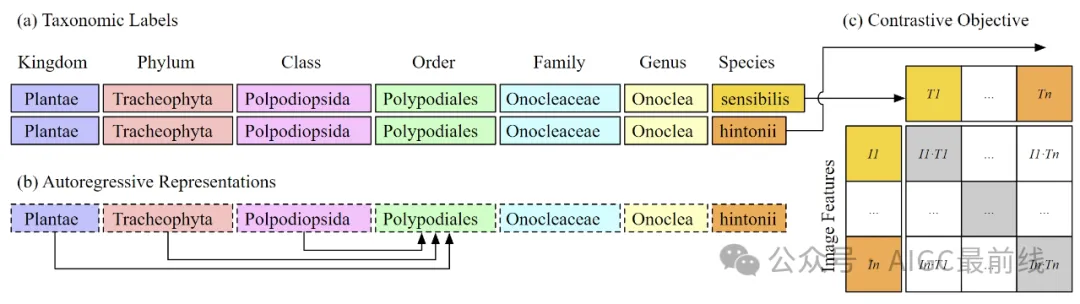
圖1:使用CLIP目標(c)來訓練一個ViT-B/16模型,該模型使用超過45萬個不同的類標簽,所有這些標簽都來自生命之樹的分類標簽(a)。由于文本編碼器是一個自回歸語言模型,因此排序表示僅能依賴于高級別的分類,如類、門和界(b)。這自然導致了標簽的層次表示,有助于視覺編碼器學習更符合生命之樹的圖像表示。
自然界的圖像是生物信息的超豐富來源。有許多計算方法和工具,特別是計算機視覺,可以從圖像中提取信息。但是,現有的方法是針對特定任務的定制模型,并且不能適應或擴展到新的問題、情境和數據集。
近日,研究人員開發了第一個大規模多模態模型,BioCLIP,用于圖像上的一般生物學問題。利用生物學作為計算機視覺應用領域的獨特特性:
- 植物、動物和真菌圖像的豐富性和多樣性;
- 豐富的結構化生物知識的可用性。
該工作入選CVPR 2024 Oral。作者發布了一個大規模、多樣化的生物圖像數據集:TreeOfLife-10M,訓練了BioCLIP,并在多樣化的細粒度生物分類任務上對該方法進行了嚴格的基準測試,發現BioCLIP始終顯著優于現有基線,絕對值提高了17%到20%。內在評估進一步揭示了BioCLIP已經學習到符合生命之樹的層次化表示,從而突顯了其強大的泛化能力。
實驗
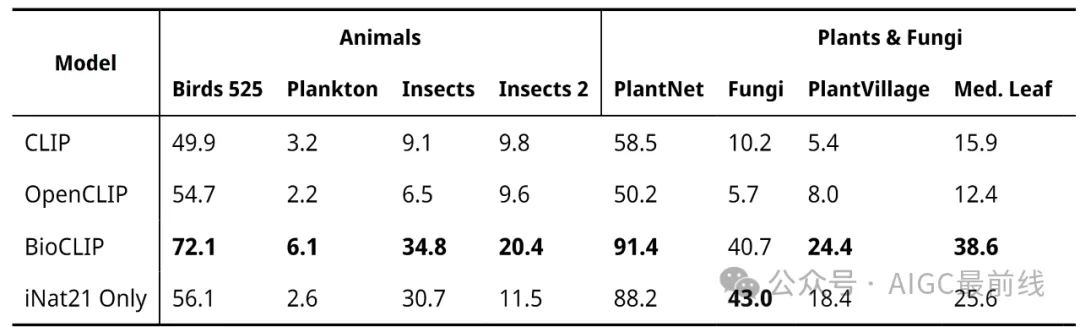
在各種各樣的與生物學相關的分類任務上,作者評估了BioCLIP和三個基準模型,包括CLIP、OpenCLIP,以及僅使用與BioCLIP相同過程但僅在iNat21上訓練的iNat-only模型。作者對所有模型進行了零樣本分類,并在驗證集上報告準確率。加粗表示每個任務的最佳性能。
實驗結果表明,BioCLIP優于通用領域的基準模型以及iNat-only模型。
內在評估
為什么BioCLIP效果這么好?作者進行了內在評估,以了解BioCLIP學習到的表示。使用T-SNE在二維平面上繪制了iNat21驗證集中10萬個未見過的圖像的BioCLIP和CLIP的表示,并根據它們的類別對點進行著色。在下圖中,(B)表示BioCLIP,(O)表示OpenAI的CLIP。
在類群等較高級別上,CLIP和BioCLIP都有良好的分離性,但您可以看到BioCLIP的表示更加細粒化,并包含更豐富的聚類結構。在較低級別上,BioCLIP產生了更具可分辨性的特征,而CLIP的特征往往混亂且缺乏清晰的結構。這表明BioCLIP已經學習到了符合分類學層次結構的豐富特征表示,這有助于解釋其在生命之樹上強大的泛化能力。
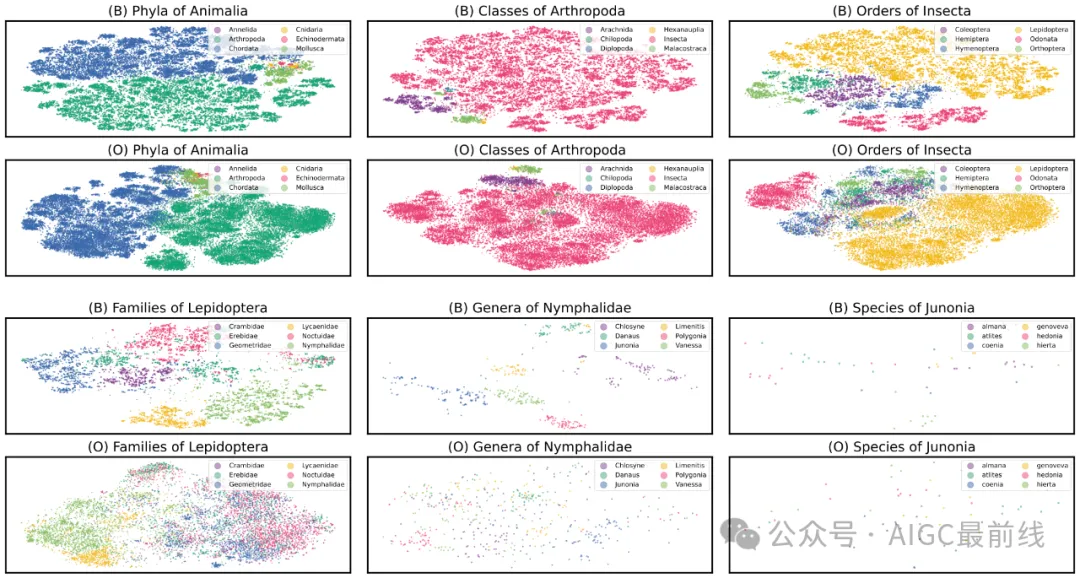
BioCLIP的表示比OpenAI的CLIP更細粒化,并且包含更豐富的聚類結構。
數據集

TreeOfLife-10M是目前可用的最大和最多樣化的生物圖像數據集。研究人員從三個來源,包括iNaturalist、BIOSCAN-1M和Encyclopedia of Life中獲取圖像,創建了一個包含1000萬張圖像的數據集,涵蓋了45萬多個物種。
參考:
@article{stevens2023bioclip, title={BioCLIP: A Vision Foundation Model for the Tree of Life}, author={Samuel Stevens and Jiaman Wu and Matthew J Thompson and Elizabeth G Campolongo and Chan Hee Song and David Edward Carlyn and Li Dong and Wasila M Dahdul and Charles Stewart and Tanya Berger-Wolf and Wei-Lun Chao and Yu Su}, year={2023}, eprint={2311.18803}, archivePrefix={arXiv}, primaryClass={cs.CV} }
本文轉載自公眾號AIGC最前線
原文鏈接:??https://mp.weixin.qq.com/s/KgvscZ321hvB-oN2VB7yDQ???

















