AI+Science:基于飛槳的AlphaFold2,帶你入門蛋白質結構預測
1958年F.H.C. 克里克提出了生物學中重要的中心法則,DNA->RNA->蛋白質,中心法則說明,DNA可以轉錄形成RNA,RNA再翻譯成一個個氨基酸,最后組合形成蛋白質。
通過中心法則不難看出,如果把DNA比喻為進行工業生產的設計藍圖,那么蛋白質就像實現這個藍圖的工具,所以說蛋白質是一切生命活動的基礎,它幾乎參與了所有的生物學過程,如遺傳、發育、繁殖等等。對蛋白質進行深入地研究,能讓我們從更深層次詮釋生命體的構成和運作變化規律,進而全面揭示生命運行、發展的機制,激發生物科學、藥物研發、合成生物學、酶科學等領域的發展。
因探究生物體內各種蛋白質的功能及其機制等是目前蛋白質研究的主要內容,同時也是后基因組時代生命科學領域的主要研究熱點之一。蛋白質的功能很大程度上取決于蛋白質的結構,因此如何破解蛋白質的三維結構成為了科學家研究的重點。
AlphaFold2的誕生
近些年來,隨著人工智能技術的發展,深度學習等相關技術也被應用在蛋白質結構預測領域。2018年的CASP 13(國際權威的蛋白質結構預測競賽,每2年舉辦一次)上,谷歌DeepMind團隊的AlphaFold拿下了70多分,打敗眾多研究團隊,取得人工組第一,在該領域取得了里程碑式的進展。在2020年的CASP 14上,谷歌DeepMind團隊的AlphaFold2以驚人的92.4分登頂第一[1],這一結果也被認為是基本解決了“困擾了生物學家50年”的問題,獲得重大突破。92.4分,指的是對競賽目標蛋白的預測精度GDT_TS分數達到92.4,一般認為該分數超過90分,基本可以替代實驗方式啦,這也意味著AlphaFold2預測的結果與實驗得到的蛋白質結構基本一致。
2021年7月15日, DeepMind團隊在國際頂級期刊《Nature》上發表論文,詳細描述了AlphaFold2的設計思路,并提供了可供運行的基于JAX的模型和代碼[2]。考慮到JAX受眾偏向專業的AI科學計算研究人員,且飛槳社區尚沒有蛋白質結構預測相關的開源項目,百度螺旋槳PaddleHelix生物計算團隊,基于飛槳深度學習框架,復現了AlphaFold2模型,提供給廣大飛槳開發者使用,幫助大家快速入門蛋白質結構預測。
https://github.com/PaddlePaddle/PaddleHelix/tree/dev/apps/paddlefold
AlphaFold2算法的設計思路
AlphaFold2通過獨特的神經網絡和訓練過程設計,第一次端到端地學習蛋白質結構。整個算法框架通過協同學習蛋白質的多序列比對(MSA)和氨基酸對(pairwise)的表征,將蛋白質序列的進化信息、蛋白質結構的物理和幾何約束信息結合到深度學習網絡中。我們將從數據預處理、Evoformer和Structure Module三個模塊分析AlphaFold2算法的設計思想。
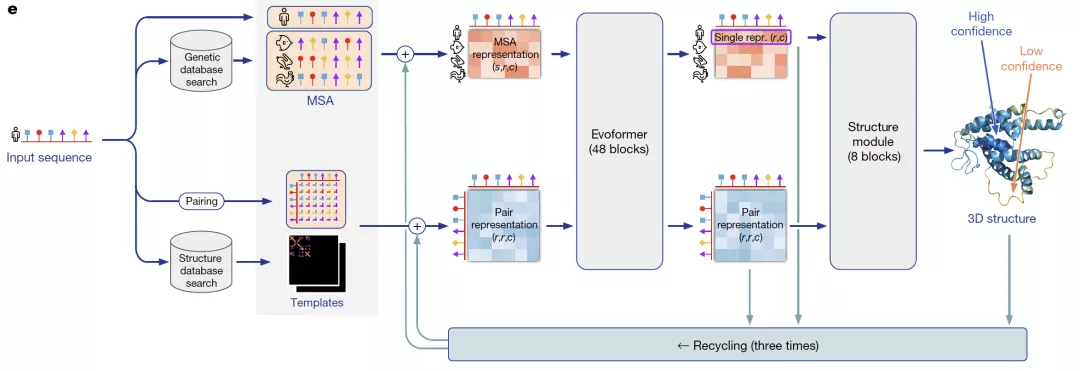
來自:AlphaFold2論文
數據處理
預測蛋白結構時,AlphaFold2會利用氨基酸序列信息在蛋白質庫中搜索多序列比對(MSA)。MSA可以反映氨基酸序列中的保守性區域(即不容易產生突變),這些保守性區域和蛋白質的結構息息相關,比如可能被折疊在蛋白質內層,不容易和外界產生相互作用,進而不易受影響發生突變。在AlphaFold2的數據預處理中,為了減少模型運算量,會先對MSA中的序列進行聚類,取每個類別中心的序列作為main MSA特征。除了MSA,AlphaFold2的另一個重要輸入是氨基酸對(pairwise)的特征。作為main MSA的補充,Alphafold2會隨機采樣非聚類中心的序列作為extra MSA輸入一個4層的網絡提取pairwise特征,然后和模版提取的pairwise特征相加后得到最終pairwise特征。main MSA特征和pairwise特征通過48層Evoformer進行表征融合。
Evoformer
Evoformer網絡的設計動機是想利用Self-Attention機制學習蛋白質的三角幾何約束信息,同時讓MSA表征帶來的共進化信息和pairwise表征的結構約束信息相互影響,使得模型能直接推理出空間信息和進化信息的聯系。
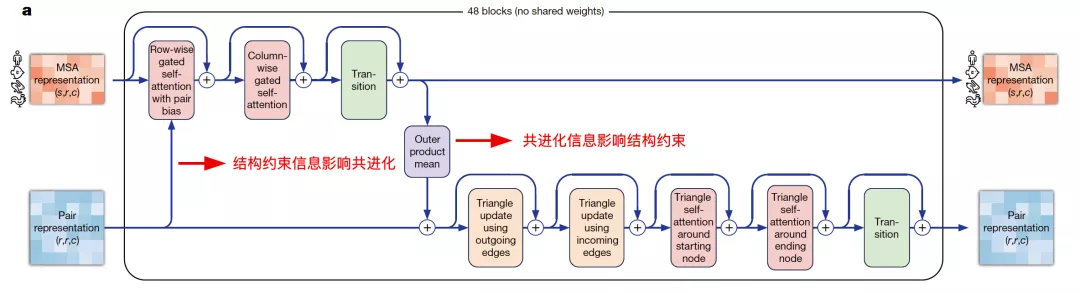
來自:AlphaFold2論文
Structure Module
Structure Module承擔著把Evoformer得到的表征解碼成蛋白質中每個重原子(C,N,O,S)坐標的任務。為了簡化從神經網絡預測值到原子坐標的轉換,AlphaFold2結合蛋白質中20類氨基酸的結構特性,將重原子分成不同二面角轉角決定的組,這樣就可以根據給定的起始位置,利用二面角和氨基酸已知的鍵長鍵角信息解碼出原子坐標。這種結構編碼方法相比直接預測坐標(x,y,z)大大降低了神經網絡的預測空間,使得端到端結構學習成為可能。

賴氨酸的轉角編碼方式示例:藍色平面(C,Cα,Cβ)確定后,根據預測的藍色-紫色平面的二面角χ1和已知的C-C鍵長,Cγ-Cβ-N鍵角即可確定Cγ的空間坐標,重復類似步驟,可以得到Cδ,Cε, N等重原子坐標。
基于飛槳框架的AlphaFold2(AF2)使用
目前已經基于飛槳框架復現了完整的AlphaFold2的inference部分,現已正式在螺旋槳PaddleHelix平臺開源:https://github.com/PaddlePaddle/PaddleHelix/tree/dev/apps/paddlefold
感興趣的小伙伴們可以安裝使用,并基于此,優化自己的蛋白結構預測模型。
1. 安裝
在requirements.txt中提供了通過pip可安裝的Python依賴項。另外,(基于飛槳框架的AF2還依賴于兩個只能通過conda安裝的 工具包:openmm==7.5.1和 pdbfixer。為了得到多序列比對MSA,還需要安裝kalign, HH-suite?和 jackhmmer。下載數據的腳本需要aria2c。
提供一個可以設置conda環境并安裝所有依賴項的腳本setup_env。運行:
sh setup_env
conda activate paddlefold # activate the conda environment
也可以在 setup_env中更改環境名稱和CUDA版本。
2. 用法
為了運行基于飛槳框架的AF2,還需要蛋白序列數據庫和模型參數。基于飛槳框架的AF2使用和AlphaFold2一樣的模型參數。
你可以使用腳本scripts/download_all_data.sh來下載和設置所有數據庫和模型參數。
- 運行:
scripts/download_all_data.sh <DOWNLOAD_DIR>
將下載完整的數據庫。完整數據庫的總下載大小約為415 GB,解壓后的總大小為2.2 TB。
- 運行:
scripts/download_all_data.sh <DOWNLOAD_DIR> reduced_dbs
將下載一個減少版本的數據庫,可以用于在reduced_ dbs的設置下運行。減少的數據庫的總下載大小約為190GB,解壓縮后的總下載大小約為530GB。
3. 運行基于飛槳框架的AF2進行推理
要使用DeepMind已經訓練好的參數對一個序列或多個序列進行推理,運行例如:
fasta_file="target.fasta" # path to the target protein
model_name="model_1" # the alphafold model name
DATA_DIR="data" # path to the databases
OUTPUT_DIR="paddlefold_output" # path to save the outputs
python3 run_paddlefold.py \
--fasta_paths=${fasta_file} \
--data_dir=${DATA_DIR} \
--small_bfd_database_path=${DATA_DIR}/small_bfd/bfd-first_non_consensus_sequences.fasta \
--uniref90_database_path=${DATA_DIR}/uniref90/uniref90.fasta \
--mgnify_database_path=${DATA_DIR}/mgnify/mgy_clusters_2018_12.fa \
--pdb70_database_path=${DATA_DIR}/pdb70/pdb70 \
--template_mmcif_dir=${DATA_DIR}/pdb_mmcif/mmcif_files \
--obsolete_pdbs_path=${DATA_DIR}/pdb_mmcif/obsolete.dat \
--max_template_date=2020-05-14 \
--model_names=${model_name} \
--output_dir=${OUTPUT_DIR} \
--preset='reduced_dbs' \
--jackhmmer_binary_path /opt/conda/envs/paddlefold/bin/jackhmmer \
--hhblits_binary_path /opt/conda/envs/paddlefold/bin/hhblits \
--hhsearch_binary_path /opt/conda/envs/paddlefold/bin/hhsearch \
--kalign_binary_path /opt/conda/envs/paddlefold/bin/kalign \
--random_seed=0
你可以使用python3 run_paddlefold.py -h來查找參數的描述。
保留與AlphaFold2相同的輸出,輸出將位于output_dir的子文件夾中。它們包括計算的MSAs、模型預測的蛋白結構、OpenMM優化后的結構、模型打分排序、原始模型輸出、預測元數據和模型運行計時。output_dir目錄將具有以下結構:
<target_name>/
features.pkl
ranked_{0,1,2,3,4}.pdb
ranking_debug.json
relaxed_model_{1,2,3,4,5}.pdb
result_model_{1,2,3,4,5}.pkl
timings.json
unrelaxed_model_{1,2,3,4,5}.pdb
msas/
bfd_uniclust_hits.a3m
mgnify_hits.sto
uniref90_hits.sto
每個輸出文件的內容如下:
- features.pkl
一個 pickle 文件,其中包含模型用于生成結構的輸入特性 NumPy 數組。
- unrelaxed_model_*.pdb
一個PDB 格式的文本文件,其中包含預測的結構,與模型輸出的結構完全一樣。
- relaxed_model_*.pdb
一個PDB格式的文本文件,是調用OpenMM得到的優化結構,修復了模型預測結構中的沖突,并添加H原子的坐標位置。
- ranked_*.pdb
一個 PDB 格式的文本文件,是對OpenMM得到的優化結構按照模型置信度的重新排序。這里使用預測的LDDT分數 (pLDDT)作為置信度評估。
- ranking_debug.json
一個JSON格式的文本文件,包含用于執行模型排名的pLDDT值及其對應的模型名稱。
- timings.json
一個JSON格式的文本文件,包含運行AlphaFold2模型的每個部分所花費的時間。
- msas/
該目錄中包含不同MSA搜索工具的輸出文件。
- result_model_*.pkl
一個pickle文件,其中包含一個由模型直接生成的各種 NumPy 數組的字典,除了結構模塊的輸出外,還包括輔助輸出。
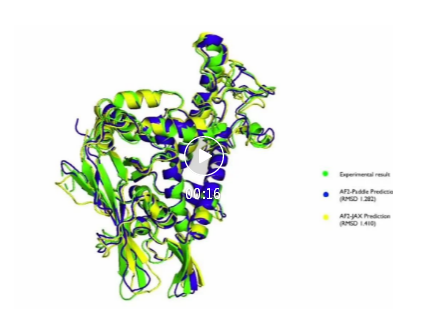
最后,可以使用pymol[3]等工具對預測結構和實驗結構對齊。值得說明的是,由于輸入特征存在采樣操作,基于飛槳框架復現的AlphaFold2和JAX版本的預測結構可能會有略微差異,有時候會和實驗結構更接近,也可能差別稍大。
近期開發計劃
AlphaFold2雖然在單體蛋白上表現優異,但對復合體,預測的準確度還有待提升。為此,DeepMind團隊上線了AlphaFold-Multimer模型,一款針對復合物進行重新訓練的神經網絡模型,希望能發動飛槳社區開發者們的積極性,一起開發優化基于AlphaFold-Multimer的模型,之后也開源貢獻到飛槳平臺,讓更廣大的生信領域研究者們使用基于飛槳框架完全自主可控的蛋白結構預測模型。
本文來源于飛槳PaddlePaddle