深度學習精確預測RNA,需訓練18種已知結構:研究登上Science封面
我們距離精確預測 DNA 結構還遠嗎?
目前,使用人工智能預測化合物分子結構是一個火熱的研究課題,DeepMind 蛋白質結構預測工具 AlphaFold2 證明了這一點。但應看到,實現分子結構準確預測的背后需要龐大的數據集。斯坦福大學的一項研究打破了這一限制,他們提出的機器學習方法僅使用很少的數據即實現了準確的 RNA 結構預測。
確定生物分子的 3D 形狀是現代生物學和醫學發現中最困難的問題之一。許多公司和研究機構花費數百萬美元來確定分子結構,卻也常常無果。
來自斯坦福大學的研究團隊利用機器學習的方法解決了這個難題。在計算機科學系副教授 Ron Dror 的指導下,斯坦福大學博士生 Stephan Eismann 和 Raphael Townshend 巧妙地使用機器學習技術開發了一種通過計算預測生物分子準確結構的方法。并且即使僅從少數已知結構中學習,他們的方法也能成功,使其適用于結構最難通過實驗確定的分子類型。
8 月 27 日,該團隊與斯坦福大學生物化學系副教授 Rhiju Das 合作的研究論文在《Science》上發表并登上封面。

論文地址:http://science.sciencemag.org/content/373/6558/1047
在此之前,去年 12 月該團隊的一篇研究論文已經登上了生物醫學期刊《Proteins》。

論文地址:https://onlinelibrary.wiley.com/doi/10.1002/prot.26033
在《Proteins》的論文中,研究團隊介紹說:該研究建立的神經網絡架構從包含數萬個原子的分子結構中進行端到端的學習,其中涉及基于點的原子表示、旋轉和平移的等變性、局部卷積和分層子采樣操作。
兩篇論文的主要作者 Townshend 說:「結構生物學是對分子形狀的研究,結構決定功能。」該團隊設計的算法不僅可以預測準確的分子結構,還能夠解釋不同分子的工作原理,該方法將適用于基礎生物學研究、藥物研發等。具體來講,團隊成員 Eismann 以蛋白質舉例說明:「蛋白質是執行各種功能的分子機器。為了執行它們的功能,蛋白質通常會與其他蛋白質結合。如果已知一對蛋白質與疾病有關,并且知道它們在三維條件下如何相互作用,醫學上就可以嘗試用一種藥物非常具體地針對這種相互作用。」
該研究的方法已經在蛋白質復合物和 RNA 分子方面取得了成功。正如研究團隊成員 Dror 所說:「機器學習近來取得的大多數進展都需要大量數據進行訓練。而該研究的方法在訓練數據很少的情況下取得成功的事實意味著:相關方法可以解決許多數據稀缺的領域中未解決的問題」,因此該方法可能具有巨大潛力。
使用少量數據實現 RNA 準確結構預測
RNA 分子的 3D 結構對 RNA 分子發揮自身功能至關重要,在藥物發現中也很有意義。然而,已知的 RNA 結構很少,并且通過計算來預測 RNA 結構極具挑戰性。
而斯坦福大學的這項研究使用機器學習(ML)的方法,只使用 18 種已知的 RNA 結構進行訓練,就能夠識別出準確的結構模型,同時無需這些結構模型的定義特性。通過這種機器學習方法得到的評分函數——原子旋轉等變評分器(Atomic Rotationally Equivariant Scorer, ARES)顯著優于以往方法。
下圖為訓練集中的 18 種 RNA 結構圖示。

具體地,為了訓練 ARES,研究者使用了 1994 年至 2006 年之間已發表的 18 個 RNA 分子,并利用 Rosetta FARFAR2 采樣方法生成了每個 RNA 的 1000 個結構模型,同時沒有使用任何已知結構。接著,他們優化了 ARES 神經網絡的參數,使其輸出盡可能匹配每個模型對應結構的均方根誤差(RMSD)。
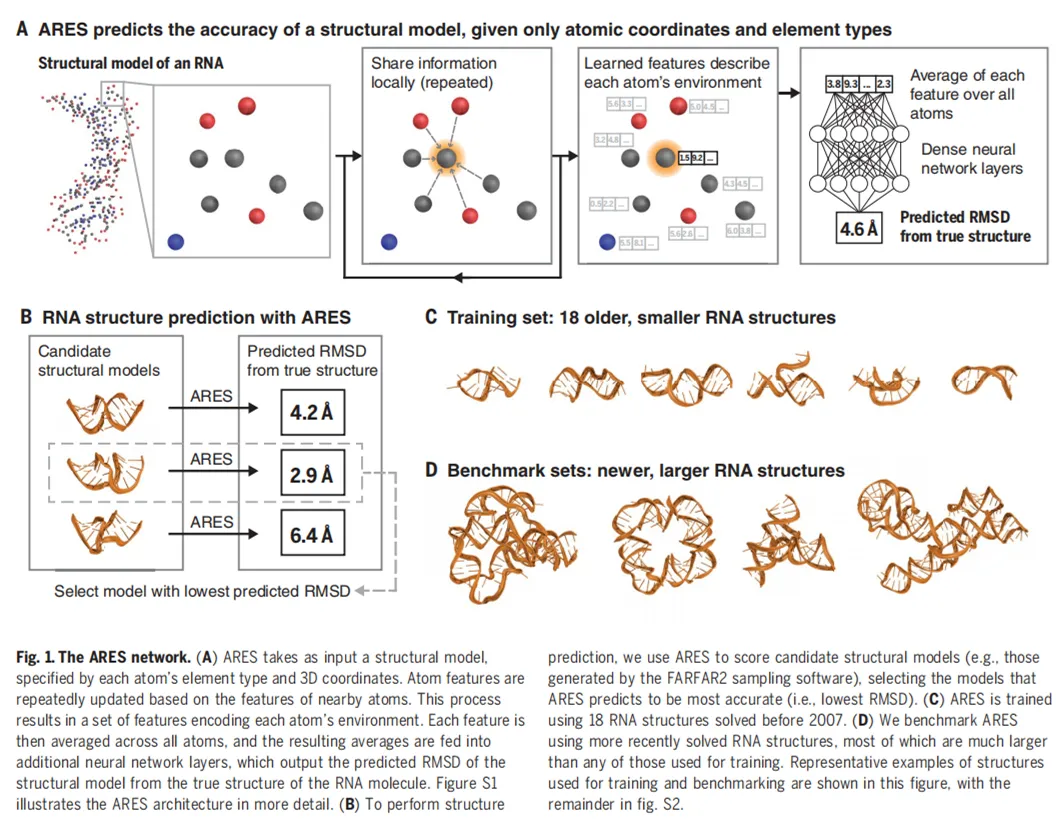
下圖 1 為 ARES 網絡,其中 A 表示:僅給出原子坐標和元素類型,ARES 即可預測結構模型的準確率;B 表示利用 ARES 的 RNA 結構預測;C 表示包含 18 種已有小型 RNA 結構的訓練集;D 表示包含新的、更大 RNA 結構的基準集。
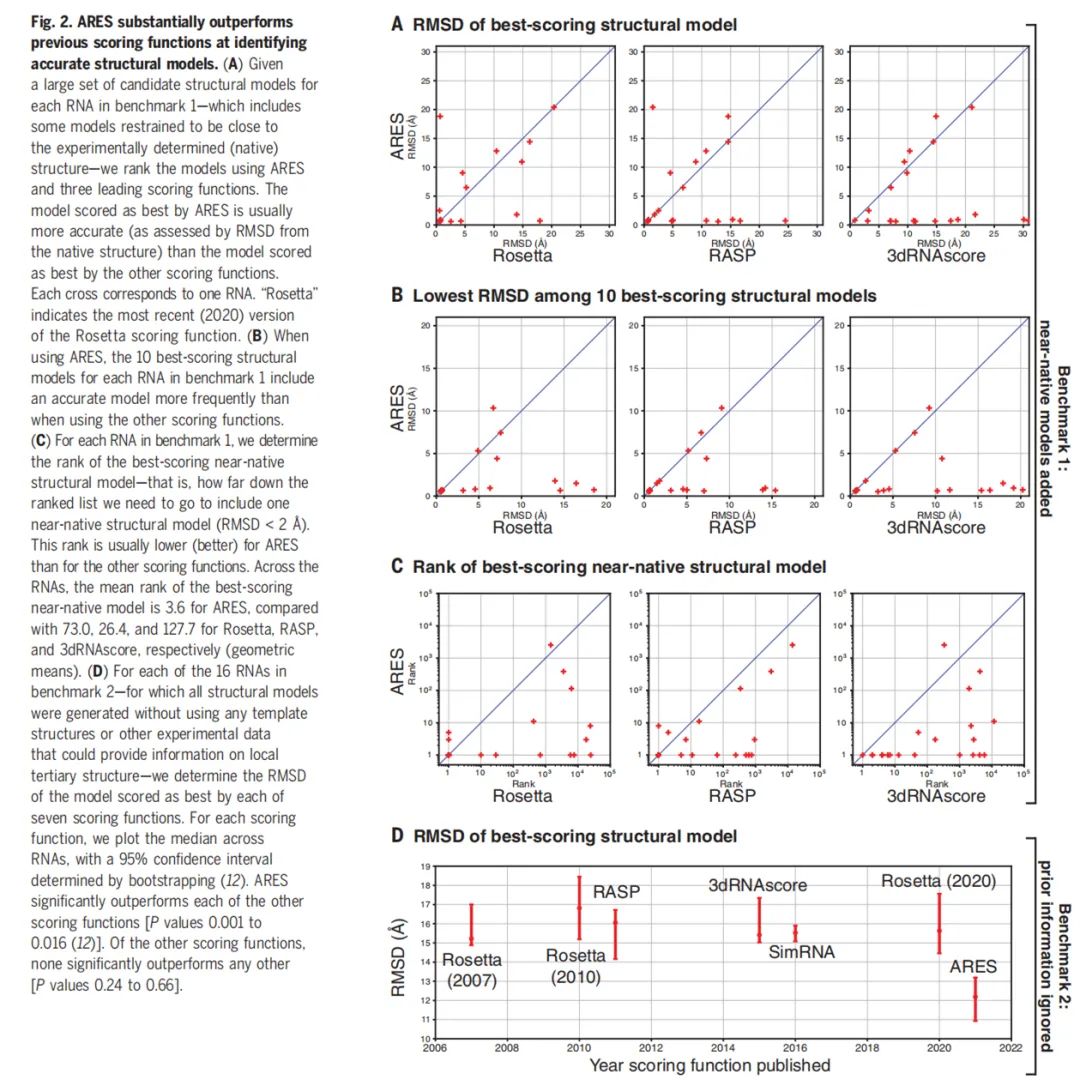
下圖 2 展示了 ARES 顯著優于以往識別準確結構模型的評分函數。A 表示評分最佳結構模型的 RMSD;B 表示 10 個評分最佳結構模型中最低的 RMSD;C 表示無限接近評分最佳結構模型的排名;D 表示 2007 年以來評分最佳結構模型的 RMSD。
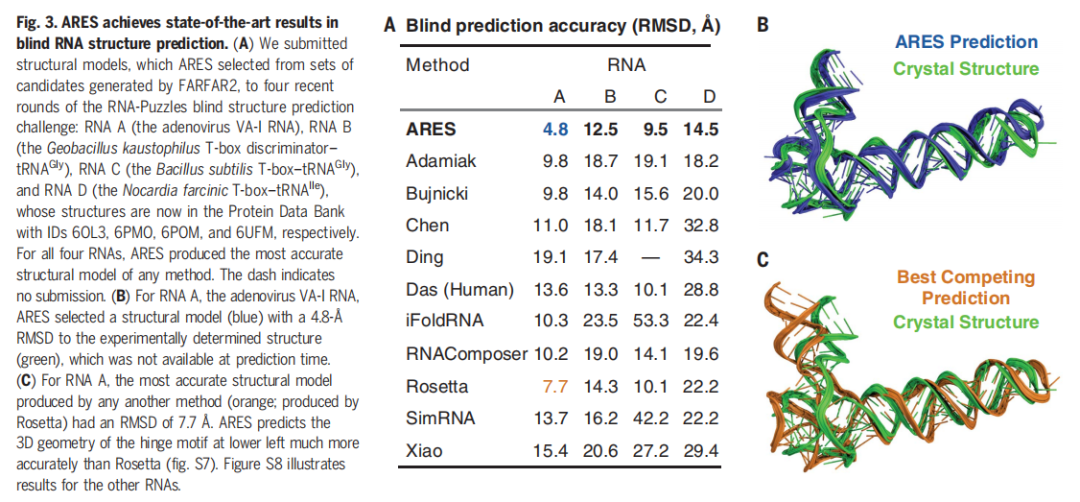
ARES 在社區范圍內的 Blind RNA 結構預測挑戰賽「RNA Puzzles」中實現了 SOTA 結果。如下圖 3 所示,A 表示 ARES 與其他方法的 Blind 預測準確率結果對比;B 表示 ARES 預測的晶體結構;C 表示其他方法實現的最佳晶體結構預測。
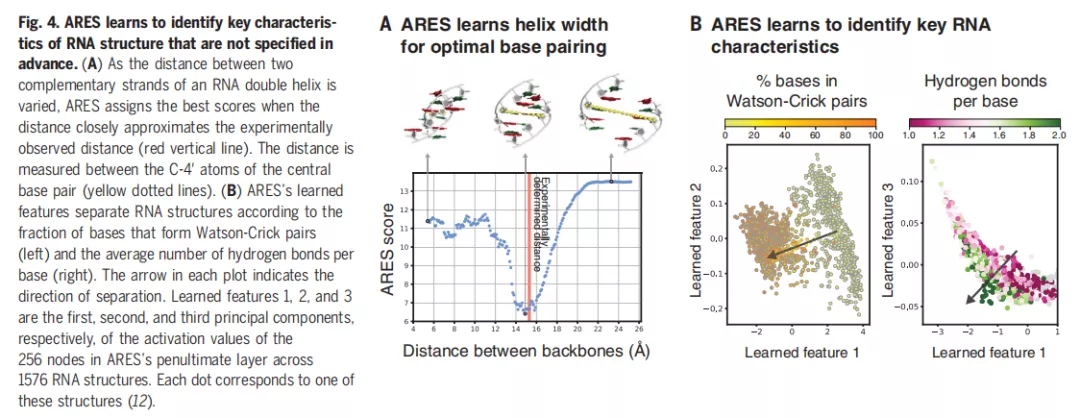
此外,ARES 還能學習識別事先沒有明確說明的 RNA 結構的關鍵特征。下圖 4A 表示 ARES 學習螺旋寬度以實現最優的堿基配對;4B 表示學習識別關鍵的 RNA 特征。
就其優勢而言,斯坦福大學的方法可以基于少量數據進行有效學習,克服了標準深度神經網絡的主要限制。并且該方法僅使用原子坐標作為輸入,不包含特定的 RNA 信息,因此適用于解決結構生物學、化學、材料科學等領域的各種問題。
作者簡介
共同一作 Raphael Townshend 是分子和藥物設計 AI 初創公司 Atomic AI 的 CEO。目前,他在斯坦福 AI 實驗室攻讀博士,研究興趣包括機器學習、結構生物學、高性能計算和計算機視覺。
通訊作者之一 Ron Dror 是斯坦福大學計算機科學系副教授,也是斯坦福 AI 實驗室所屬一個研究小組的負責人,專注于使用計算機技術解決生物學和藥物發現的基礎問題。作為高性能計算、機器學習、圖像分析、結構生物學和藥物設計等領域的專家,他還教授計算機生物學和機器學習,并為技術和制藥企業提供咨詢。