醫療圖像分割中的深度學習方法
譯文
譯者 | 布加迪
審校 | 重樓
深度學習大大提高了醫療圖像分割的準確度和效率,本文將探討常用技術及其應用。
采用深度學習技術已經使醫療成像發生了革命性的變化。使用機器學習的這一分支開創了醫療圖像分割精度和高效的新時代,而醫療圖像分割是現代醫療保健診斷和治療計劃的一個核心分析過程。通過利用神經網絡,深度學習算法能夠以前所未有的精度檢測醫療圖像中的異常。
這項技術突破有助于重塑我們對待醫療圖像分析的范式。從改善早期疾病檢測到促進個性化治療策略,醫療圖像分割中的深度學習正在為更有針對性、更有效的患者護理鋪平道路。我們在本文中將深入研究深度學習為醫療圖像分割領域帶來的變革性方法,探索這些先進的算法如何推動醫療成像發展、乃至推動醫療領域本身發展。
醫療圖像分割簡介
醫療圖像分割指將圖像分割成不同的區域。每個區域代表一個特定的結構或特征,比如器官或腫瘤。這個過程對于解讀和分析醫療圖像很重要。它可以幫助醫生更準確地診斷疾病。分割有助于規劃治療和跟蹤病人的病情變化。
用于圖像分割的常見深度學習架構
不妨先看看將深度學習用于圖像分割的幾種常見架構:
1.U-Net
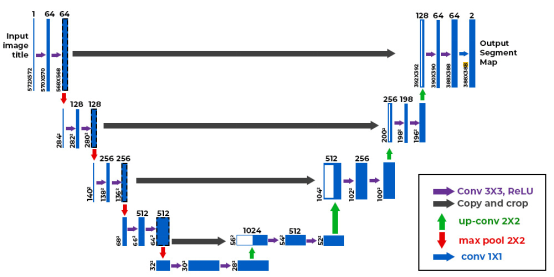
U-Net有一個U形,有用于上下文的編碼器和用于精確定位的解碼器。U-Net中的跳過連接保留了編碼器層和解碼器層的重要細節。U-Net有助于在MRI和CT掃描圖中分割器官、腦腫瘤、肺結節及其他關鍵結構。
2.全卷積網絡(FCN)
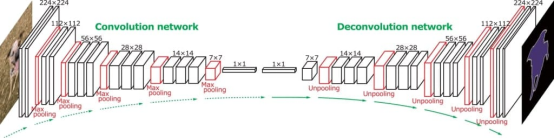
FCN在整個網絡中使用卷積層,而不是使用完全連接的層。這使模型能夠生成密集的分割圖。FCN借助上采樣技術保持輸入圖像的空間維度。它們有助于對每個像素單獨進行分類。比如說,它們有助于在MRI掃描圖中發現腦腫瘤,并在CT圖像中顯示肝臟的位置。
3.SegNet
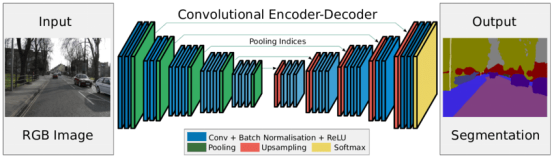
SegNet兼顧了性能和計算效率。其編碼器-解碼器設計先減小圖像尺寸,然后再將其放大以創建詳細的分割圖。SegNet在編碼期間存儲最大池索引,并在解碼期間重用它們以提高準確性。它被用于分割視網膜血管、X光下的肺葉及效率很重要的其他結構。
4.DeepLab
DeepLab在保持空間分辨率的同時,使用空洞卷積來擴展接受域。ASPP模塊捕獲不同尺度的特征。這有助于模型處理分辨率各異的圖像。DeepLab用于處理發現MRI掃描圖中的腦腫瘤、肝臟病變和心臟細節等任務。

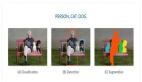
示例:U-Net肺腫瘤分割
現在不妨看一個使用U-Net模型逐步分割肺腫瘤的例子。
1.掛載Google Drive
首先我們將掛載Google Drive,以訪問存儲在其中的文件。
from google.colab import drive
drive.mount('/content/drive')2.定義文件夾路徑
現在我們為Google Drive中含有圖像和標簽的文件夾設置路徑。
# Define paths to the folders in Google Drive
image_folder_path = '/content/drive/My Drive/Dataset/Lung dataset'
label_folder_path = '/content/drive/My Drive/Dataset/Ground truth'3.收集PNG文件
接下來,定義一個函數來收集和排序指定文件夾中的所有PNG文件路徑。
# Function to collect PNG images from a folder
def collect_png_from_folder(folder_path):
png_files = []
for root, _, files in os.walk(folder_path):
for file in files:
if file.endswith(".png"):
png_files.append(os.path.join(root, file))
return sorted(png_files)4.加載和預處理數據集
接下來我們將定義一個函數,從各自的文件夾中加載和預處理圖像和標簽。該函數確保圖像和標簽正確匹配并調整大小。
# Function to load images and labels directly
def load_images_and_labels(image_folder_path, label_folder_path, target_size=(256, 256), filter_size=3):
# Collect file paths
image_files = collect_png_from_folder(image_folder_path)
label_files = collect_png_from_folder(label_folder_path)
# Ensure images and labels are sorted and match in number
if len(image_files) != len(label_files):
raise ValueError("Number of images and labels do not match.")
# Load images
def load_image(image_path):
image = cv2.imread(image_path, cv2.IMREAD_GRAYSCALE)
if image is None:
raise ValueError(f"Unable to load image: {image_path}")
image = cv2.resize(image, target_size)
image = cv2.medianBlur(image, filter_size)
return image.astype('float32') / 255.0
# Load labels
def load_label(label_path):
label = cv2.imread(label_path, cv2.IMREAD_COLOR)
if label is None:
raise ValueError(f"Unable to load label image: {label_path}")
return cv2.resize(label, target_size)
images = np.array([load_image(path) for path in image_files])
labels = np.array([load_label(path) for path in label_files])
return images, labels5.顯示圖像和標簽
現在我們將定義一個函數,并排顯示指定數量的圖像及其相應的標簽。使用前面定義的函數來加載圖像和標簽,然后顯示幾個示例以進行可視化。藍色點代表腫瘤標記。
# Function to display images and labels
def display_images_and_labels(images, labels, num_samples=5):
num_samples = min(num_samples, len(images))
plt.figure(figsize=(15, 3 * num_samples))
for i in range(num_samples):
plt.subplot(num_samples, 2, 2 * i + 1)
plt.title(f'Image {i + 1}')
plt.imshow(images[i], cmap='gray')
plt.axis('off')
plt.subplot(num_samples, 2, 2 * i + 2)
plt.title(f'Label {i + 1}')
plt.imshow(labels[i])
plt.axis('off')
plt.tight_layout()
plt.show()
# Load images and labels
images, labels = load_images_and_labels(image_folder_path, label_folder_path)
# Display a few samples
display_images_and_labels(images, labels, num_samples=5)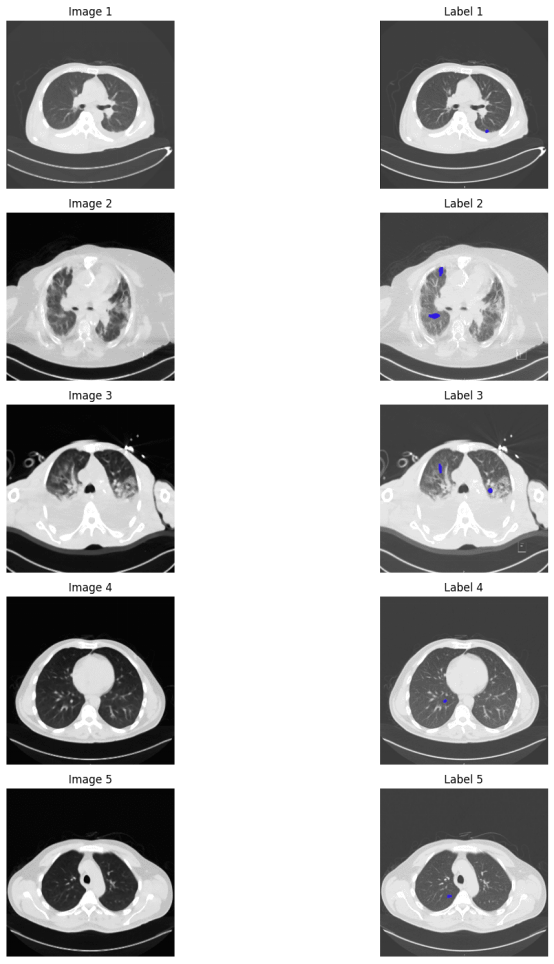
6.定義U-Net模型
現在是時候定義U-Net模型了。U-Net架構使用Adam優化器。它采用分類交叉熵作為損耗函數。準確度被用作評估指標。
# Define the U-Net model
def unet_model(input_size=(256, 256, 1), num_classes=3):
inputs = Input(input_size)
# Encoder (Downsampling Path)
c1 = Conv2D(64, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(inputs)
c1 = Dropout(0.1)(c1)
c1 = Conv2D(64, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(c1)
p1 = MaxPooling2D((2, 2))(c1)
c2 = Conv2D(128, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(p1)
c2 = Dropout(0.1)(c2)
c2 = Conv2D(128, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(c2)
p2 = MaxPooling2D((2, 2))(c2)
c3 = Conv2D(256, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(p2)
c3 = Dropout(0.2)(c3)
c3 = Conv2D(256, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(c3)
p3 = MaxPooling2D((2, 2))(c3)
c4 = Conv2D(512, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(p3)
c4 = Dropout(0.2)(c4)
c4 = Conv2D(512, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(c4)
p4 = MaxPooling2D(pool_size=(2, 2))(c4)
# Bottleneck
c5 = Conv2D(1024, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(p4)
c5 = Dropout(0.3)(c5)
c5 = Conv2D(1024, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(c5)
# Decoder (Upsampling Path)
u6 = Conv2DTranspose(512, (2, 2), strides=(2, 2), padding='same')(c5)
u6 = concatenate([u6, c4])
c6 = Conv2D(512, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(u6)
c6 = Dropout(0.2)(c6)
c6 = Conv2D(512, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(c6)
u7 = Conv2DTranspose(256, (2, 2), strides=(2, 2), padding='same')(c6)
u7 = concatenate([u7, c3])
c7 = Conv2D(256, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(u7)
c7 = Dropout(0.2)(c7)
c7 = Conv2D(256, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(c7)
u8 = Conv2DTranspose(128, (2, 2), strides=(2, 2), padding='same')(c7)
u8 = concatenate([u8, c2])
c8 = Conv2D(128, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(u8)
c8 = Dropout(0.1)(c8)
c8 = Conv2D(128, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(c8)
u9 = Conv2DTranspose(64, (2, 2), strides=(2, 2), padding='same')(c8)
u9 = concatenate([u9, c1], axis=3)
c9 = Conv2D(64, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(u9)
c9 = Dropout(0.1)(c9)
c9 = Conv2D(64, (3, 3), activation='relu', kernel_initializer='he_normal', padding='same')(c9)
# Output layer
outputs = Conv2D(num_classes, (1, 1), activation='softmax')(c9)
model = Model(inputs=[inputs], outputs=[outputs])
# Compile the model
model.compile(optimizer='adam',
loss='categorical_crossentropy',
metrics=['accuracy'])
return model7.訓練U-Net模型
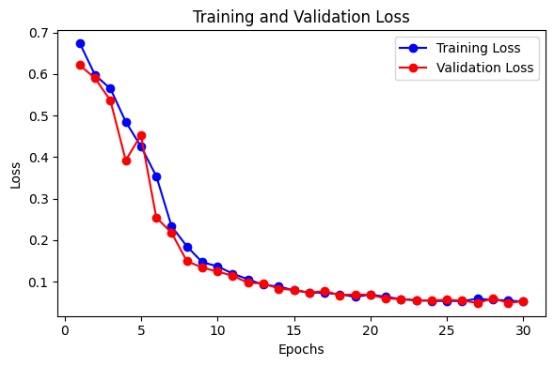
這里我們將訓練U-Net模型,并將其保存到一個文件中。訓練和驗證在輪次期間的準確性和損失被繪制成圖,以直觀顯示模型的性能。該模型可用于對新數據進行測試。
from sklearn.model_selection import train_test_split
# Split the data into training, validation, test sets
X_train, X_temp, y_train, y_temp = train_test_split(X, Y, test_size=0.4, random_state=42)
X_val, X_test, y_val, y_test = train_test_split(X_temp, y_temp, test_size=0.5, random_state=42)
# Define EarlyStopping callback
early_stopping = EarlyStopping(monitor='val_loss', patience=3, restore_best_weights=True)
# Train the model with EarlyStopping
history = model.fit(X_train, y_train,
epochs=50,
batch_size=16,
validation_data=(X_val, y_val),
callbacks=[early_stopping])
# Save the model
model.save('/content/unet_real_data.h5')
# Function to Plot Accuracy
def plot_accuracy(history):
epochs = range(1, len(history.history['accuracy']) + 1)
# Plot Training and Validation Accuracy
plt.figure(figsize=(6, 4))
plt.plot(epochs, history.history['accuracy'], 'bo-', label='Training Accuracy')
plt.plot(epochs, history.history['val_accuracy'], 'ro-', label='Validation Accuracy')
plt.title('Training and Validation Accuracy')
plt.xlabel('Epochs')
plt.ylabel('Accuracy')
plt.legend()
plt.tight_layout()
plt.show()
# Function to Plot Loss
def plot_loss(history):
epochs = range(1, len(history.history['loss']) + 1)
# Plot Training and Validation Loss
plt.figure(figsize=(6, 4))
plt.plot(epochs, history.history['loss'], 'bo-', label='Training Loss')
plt.plot(epochs, history.history['val_loss'], 'ro-', label='Validation Loss')
plt.title('Training and Validation Loss')
plt.xlabel('Epochs')
plt.ylabel('Loss')
plt.legend()
plt.tight_layout()
plt.show()
# Call the functions to plot accuracy and loss
plot_accuracy(history)
plot_loss(history)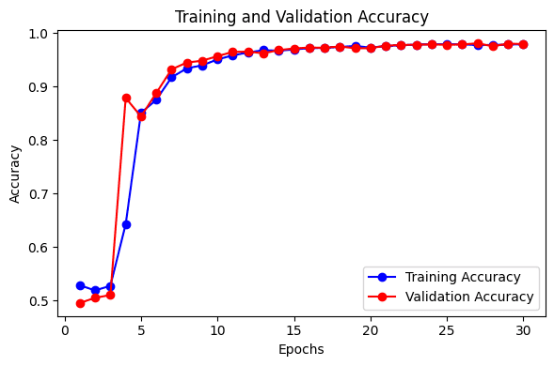
醫療圖像分割中深度學習的優點
深度學習在醫療分割中的優點有很多。以下是其中幾個重要的優點:
提高準確性:深度學習模型非常擅長準確地分割醫療圖像。它們可以發現并描繪使用舊方法可能遺漏的細小的或棘手的細節。
效率和速度:這種模型可以快速處理和分析許多圖像。它們使分割過程更快,減少了對人力工作的需求。
處理復雜數據:深度學習模型可以處理來自CT或MRI掃描圖的復雜3D圖像。它們可以處理不同類型的圖像,并適應各種成像技術。
醫療圖像分割中深度學習的挑戰
正如有優點一樣,我們也必須牢記使用這項技術面臨的挑戰。
有限的數據:始終沒有足夠的已標記醫療圖像來訓練深度學習模型。創建這些標簽很耗時,需要熟練的專家。這使得獲得足夠的數據用于訓練變得困難。
隱私問題:醫療圖像含有敏感的患者信息,因此要有嚴格的規定來保護這些數據的私密性。這意味著可能沒有那么多的數據用于研究和訓練。
可解釋性:深度學習模型可能很難理解,因此很難信任和驗證它們的結果。
結語
綜上所述,深度學習使醫療圖像分割變得更好。卷積神經網絡和Transformers等方法改進了我們分析圖像的方式,從而帶來了更準確的診斷和更好的病人護理。
原文標題:Deep Learning Approaches in Medical Image Segmentation,作者:Jayita Gulati