融合蛋白質語言模型和圖像修復模型,麻省理工與哈佛聯(lián)手提出PUPS ,實現(xiàn)單細胞級蛋白質定位 原創(chuàng)
蛋白質亞細胞定位(subcellular localization of a protein)是指蛋白質在細胞結構中具體的定位情況,這對蛋白質行使其生物學功能至關重要。舉個簡單例子,如果把細胞想象成一個龐大的企業(yè),其中細胞核、線粒體、細胞膜等對應總裁辦、發(fā)電部、門崗等不同的部門,那么只有對應的蛋白進入正確的「部門」才能使其正常工作,否則便會導致某些疾病發(fā)生,如癌癥、阿爾茲海默病。因此,精準定位蛋白質亞細胞可以說是生命科學的核心任務之一。
盡管科研界已經(jīng)對不同細胞系中的數(shù)千種蛋白質進行了空間定位分析,但到目前為止,已測量的蛋白質與細胞系組合數(shù)量還只是其中的冰山一角。比如當前最大的亞細胞定位數(shù)據(jù)集——
人類蛋白質圖譜(Human Protein Atlas,HPA),提供了 13,147 個基因編碼的蛋白質亞細胞定位(占已知人類蛋白質編碼基因的 65%),但是整個數(shù)據(jù)集包含了 37 個細胞系,而每種蛋白質最多只能在其中三株中進行測量。與此同時,主流的實驗手段很難在同一細胞中同時檢測所有蛋白質數(shù)量,這嚴重阻礙了全面分析復雜的蛋白質網(wǎng)絡,增加了實驗復雜度和誤差風險。
除此之外,蛋白質定位并非靜止不變的,它的變異性不僅體現(xiàn)在細胞系之間,甚至在同一細胞系的單個細胞間也會發(fā)生,而現(xiàn)有數(shù)據(jù)圖譜記錄的蛋白質和細胞系對僅反映了特定條件下的結果。因此,
即便是現(xiàn)有成果也很難直接套用,需要根據(jù)環(huán)境變化而對蛋白質定位進一步探索。
為了解決蛋白質亞細胞定位技術方法的局限性和生物系統(tǒng)復雜性之間的矛盾,機器學習被寄予厚望。如今已經(jīng)建模并成功應用的如基于蛋白質序列的模型、基于細胞圖像的模型等,雖然在某些方面表現(xiàn)亮眼,但不足之處也十分突出——前者忽視了細胞類型的特異性定位差異,后者則缺乏推向未知蛋白研究的泛化能力。
有鑒于此,
來自美國麻省理工學院和哈佛大學的研究團隊提出了一種結合蛋白質序列和細胞圖像來進行未知蛋白質亞細胞定位的預測框架,命名為 Predictions of Unseen Proteins’ Subcellular localization(PUPS)。PUPS 創(chuàng)新地結合了蛋白質語言模型和圖像修復模型來預測蛋白質定位,使其兼并推向未知蛋白預測的泛化能力和捕獲細胞可變性的細胞類型特定預測。實驗證明,該框架能夠準確預測訓練數(shù)據(jù)集之外新實驗中蛋白質的定位,具有極佳的泛化能力和高度的準確性,應用潛力突出。

PUPS 技術研究背景,目標及現(xiàn)有數(shù)據(jù)的局限性
研究成果以「Prediction of protein subcellular localization in single cells」為題,已發(fā)表于 Nature Methods。
研究亮點:
* 所提研究創(chuàng)新地結合了蛋白質語言模型和圖像繪制模型,利用蛋白質序列和細胞圖像進行蛋白質定位預測,彌補了過往計算模型的不足
* PUPS 能夠推廣到未知蛋白質和細胞系,從而評估細胞系之間以及細胞系內單個細胞間蛋白質定位的變異性,并識別與具有可變定位的蛋白質相關的生物過程
* 在訓練數(shù)據(jù)集之外的新實驗中,PUPS 同樣展示了其高度精確的預測能力,具有突出的應用潛力和醫(yī)學價值

論文地址:
??https://go.hyper.ai/LeaQF???
數(shù)據(jù)集地址:
數(shù)據(jù)集:以盡可能全面的數(shù)據(jù)打造可信模型
PUPS 的訓練數(shù)據(jù)集來自于人類蛋白質圖譜(Human Protein Atlas,HPA),研究團隊將第 16 版 HPA 數(shù)據(jù)匯總到第 22 版當中,以盡可能多的收集蛋白質的數(shù)據(jù),確保實驗分析的全面性。如下圖所示:
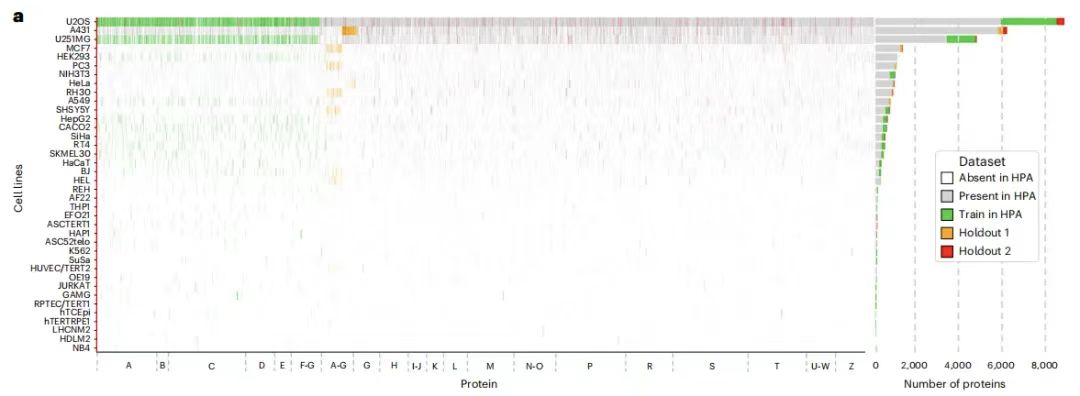
訓練集(綠色),保留集 1 (橙色),保留集 2 (紅色)
HPA 中未采用部分(灰色),HPA 中不包含部分(白色)
具體來說,訓練數(shù)據(jù)集包含 340,553 個細胞數(shù)量,蛋白質變體共 8,086 種,對應 HPA 中 37 種細胞系中的 2,801 個基因,這些基因名稱以字母 A-G 開頭。另外,訓練數(shù)據(jù)集中還額外包含了 10 個基因,包括 IHO1、IMPAD1、INKA1、ISPD、ITPRID1、KIAA1211L、KIAA1324、LRATD1、SCYL3、TSPAN6。
保留數(shù)據(jù)集分為兩部分:
一部分為保留數(shù)據(jù)集 1,包含 36,552 個細胞,蛋白質變體由 9,472 種構成,對應 3,312 個基因(含訓練集中的 2,801 個),名稱同樣以 A-G 開頭,但來自不同的細胞系,與訓練集無重疊。同時,保留數(shù)據(jù)集 1 進一步被拆分為兩個部分,用作評估集和測試集,分別包含 11,050 和 25,502 個細胞;
保留數(shù)據(jù)集 2 含有 24,007 個細胞,對應 515 個基因,其名稱以字母表所有字母開頭,即涵蓋 A-Z,蛋白質變體共 556 種,來自完全未在訓練集和保留數(shù)據(jù)集 1 中出現(xiàn)的新基因家族,可用于模型泛化能力的測試。
另需說明的是,BJ 細胞系圖像被同時保留在了訓練集和保留數(shù)據(jù)集 1 中。
在實驗之前,研究團隊對 HPA 中的圖像進行了預處理,簡單來說包含以下 5 步:
* 第一步,對每張圖像向下采樣 4 次,最終分辨率降至 0.32 μm/像素,以便減少計算量并去除高頻噪聲;
* 第二步,結合高斯模糊(σ=5)和 Otsu 閾值法從復雜背景中分離出細胞核的大致區(qū)域;
* 第三步,使用 remove_small_holes 函數(shù),移除面積小于 300 像素的孔洞,然后將圖像二值化,并去除小于 100 像素的噪聲區(qū)域;
* 第四步,計算每個細胞核的質心,并以質心為中心,裁剪出 128 x 128 像素的區(qū)域作為單個細胞的 ROI;
* 第五步,通過強度歸一化和噪聲過濾,實現(xiàn)標準化數(shù)據(jù)分布,減少通道間干擾。
模型架構:結合蛋白質序列和圖像表征預測蛋白質亞細胞定位
PUPS 模型主要由兩個部分組成,
一個用于從蛋白質的氨基酸序列中學習序列表示;另一個用于從靶細胞的標志性染色中學習圖像表示,然后結合蛋白質序列表示和圖像表示來預測蛋白質在靶細胞中的亞細胞定位。前者使模型能夠推廣到未知蛋白質預測,后者使模型具備捕獲單細胞水平的變異性,實現(xiàn)了細胞類型特異的定位預測。如下圖所示:
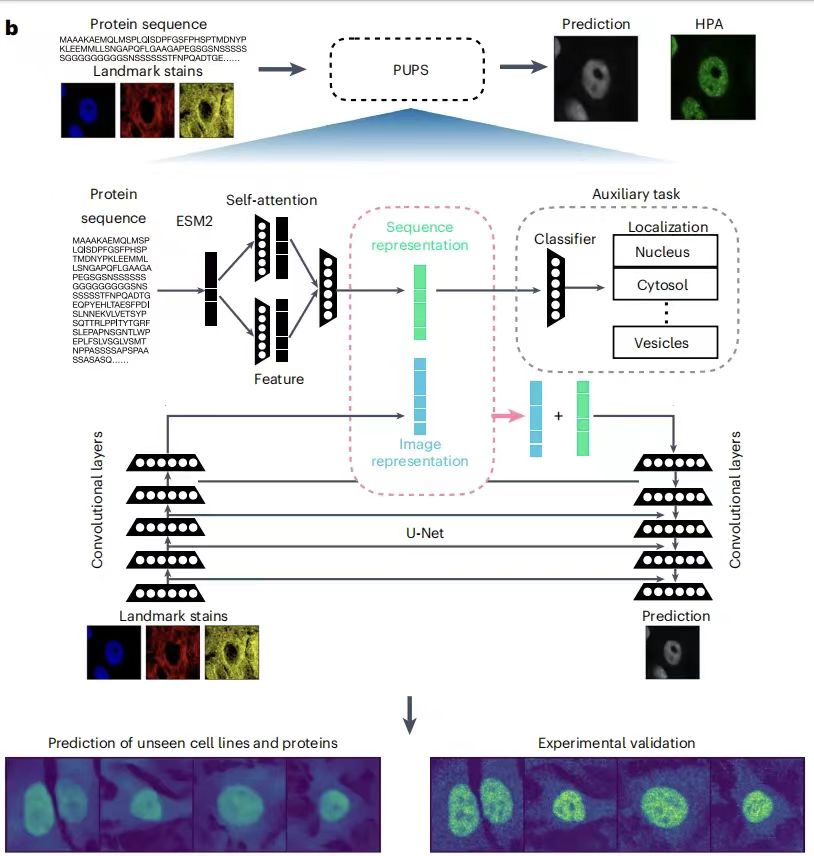
未知細胞系中未知蛋白質亞細胞定位演示
簡單來說,
PUPS 利用了預訓練的 ESM-2(Evolutionary Scale Modeling)蛋白質語言模型提取蛋白序列特征,同時用卷積神經(jīng)網(wǎng)絡學習細胞的標志性染色圖像特征,最終結合兩部分信息預測蛋白質在靶細胞中的定位。需要說明的是,模型所有部分同時進行訓練,有助于減少前置任務的分類損失,以及預測蛋白質圖像與 HPA 中實驗測量的蛋白質圖像之間的差異。所有參數(shù)使用 Adam 優(yōu)化器進行優(yōu)化,學習率為 1e-4。
蛋白質語言模型
PUPS 通過使用語言模型、自注意力層以及一個輔助預訓練任務來學習序列表征,然后根據(jù)學習到的序列表征對蛋白質定位進行分類。
具體來說,研究團隊通過將 N 端 2,000 個氨基酸序列輸入到預訓練 ESM-2 模型中,獲得特定蛋白質變體的初始表示,從而為每個氨基酸殘基生成 1,280 維向量,殘基少于 2,000 的變體采用零填充。這種序列長度截斷是為了避免對序列長度高達數(shù)萬個殘基的少數(shù)蛋白質進行偏倚預測。如下圖所示:
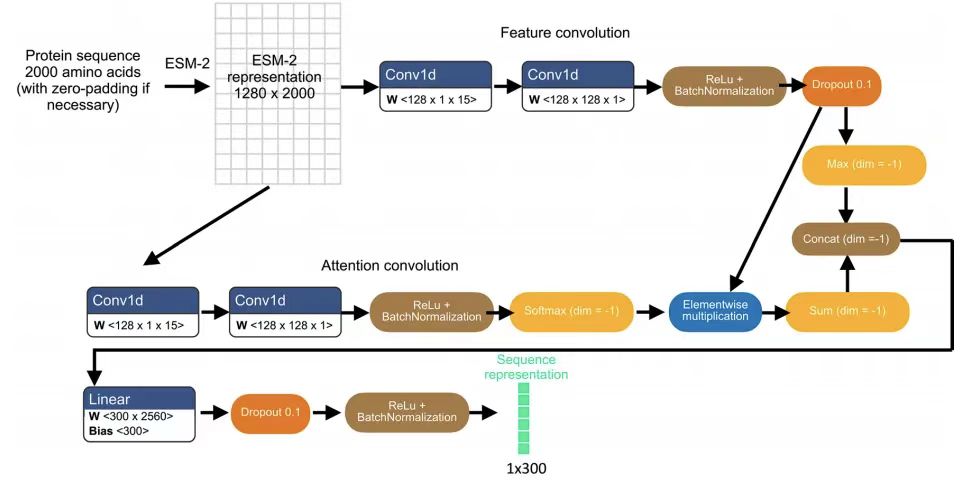
基于預訓練 ESM-2 模型與輕量注意力層的蛋白質序列表征學習模型架構
為了使 ESM-2 表征適應于蛋白質定位預測,
團隊在后續(xù)采用了可分離卷積(separable convolutions)的輕注意力層,應用于 ESM-2 表示最終獲得 300 維序列表征。這種蛋白質序列表示既用于預測定位標簽的輔助前置任務,同時也用于與圖像表示相結合的蛋白質圖像預測。前置任務將蛋白質序列表示輸入到一個全連接的神經(jīng)網(wǎng)絡層,以輸入一個 29 維向量,表示 29 個亞細胞區(qū)室定位標簽中的概率分布,然后利用 S 型激活(sigmoid activation)的二元交叉熵損失將前置任務輸出結果與 HPA 注釋的蛋白區(qū)室進行比較。
圖像繪制模型
每個細胞的圖像輸入包含了細胞核、微管和內質網(wǎng)染色這 3 個標志性染色圖像通道,其維度為 3 x 128 x 128,并以細胞核質心為中心。
圖像編碼通過 5 個可分離卷積層實現(xiàn),最終維度 16 x 16 x 512。每個卷積層之后依次連接 leakyRelu 激活,批歸一化以及 2D 最大池化層。蛋白質序列表示被拼接至細胞圖像表示的所有空間維度,隨后輸入 U-Net 圖像解碼器,為每個輸入通道學習不同權重。此外,模型中的空間維度加權機制允許圖像表征的每個空間維度以不同權重與序列表征相結合。
解碼器由 5 個可分離卷積層構成,生成 1 x 128 x 128 的圖像輸出,即對應細胞的蛋白質圖像預測。然后將類似于圖像分割 U-Net 的跳躍連接(skip connentions)添加在標志染色生成圖像表示的編碼層與同深度生成蛋白質圖像預測的解碼層之間。研究采用了均方誤差損失函數(shù)訓練模型,以最小化預測蛋白質圖像與實驗測量蛋白質圖像之間的差異。
實驗結果:實現(xiàn)單細胞級蛋白質亞細胞精準定位
為了驗證模型的可行性和有效性,研究團隊提出多項實驗進行驗證,PUPS 在多項任務中均表現(xiàn)出較好的性能,凸顯了其多模型融合的優(yōu)勢。
預測細胞系間蛋白質定位的變異性
為了評估 PUPS 在定量分析蛋白質于細胞系間定位變異性方面的性能,
研究團隊通過計算蛋白質核內比例量化定位變異性,發(fā)現(xiàn)預測值與真實數(shù)據(jù)高度相關,Holdout 1 的 pearson 相關系數(shù)為 0.794,Holdout 2 的 pearson 相關系數(shù)為 0.878。如下圖所示:
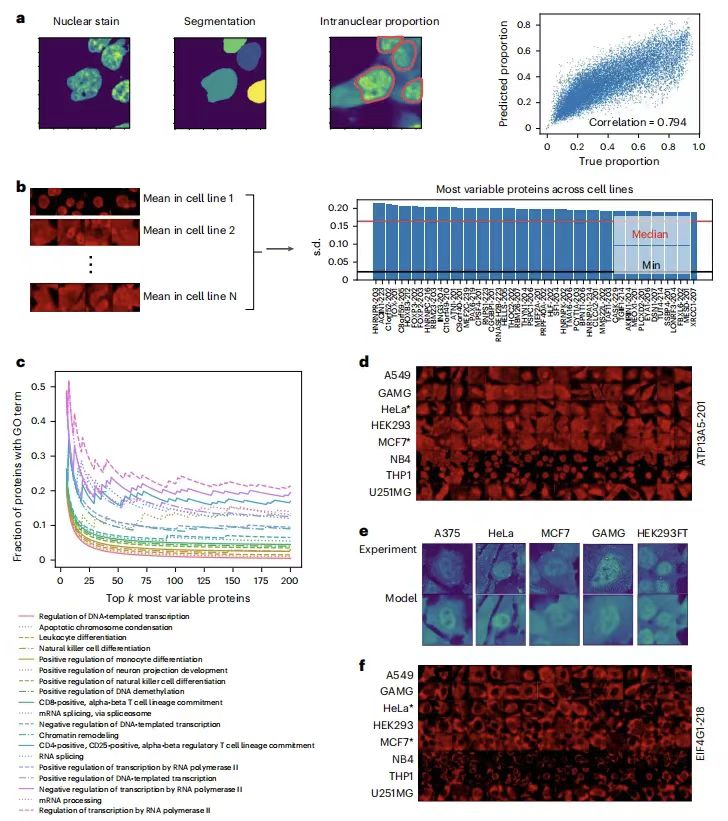
PUPS 精準預測不同細胞系間蛋白質定位的差異
隨后進一步分析顯示,細胞系間定位變化最大的蛋白質與轉錄、細胞分化和染色質調節(jié)等生物過程相關,如 ATP13A5 的實驗驗證證實了模型預測的準確性。此外,
模型通過標志性染色捕捉細胞形態(tài)差異,無需細胞系標簽即可推斷蛋白質定位的細胞系特異性,為研究蛋白質功能的細胞特異性調控提供了新方法。
預測單細胞間蛋白質定位的差異性
為了評估 PUPS 對同一細胞系內單細胞間蛋白質定位變異性的預測能力,研究團隊計算了每個細胞系中所有單細胞中蛋白質的核內比例方差,
結果發(fā)現(xiàn)每種蛋白與細胞系對的單細胞變異性預測排名與真實數(shù)據(jù)高度一致,如 Holdout 2 中前 500 個高變異對重疊率超過了 60%,并且預測的核內比例分布與實際結果一致,排除了預測誤差影響。
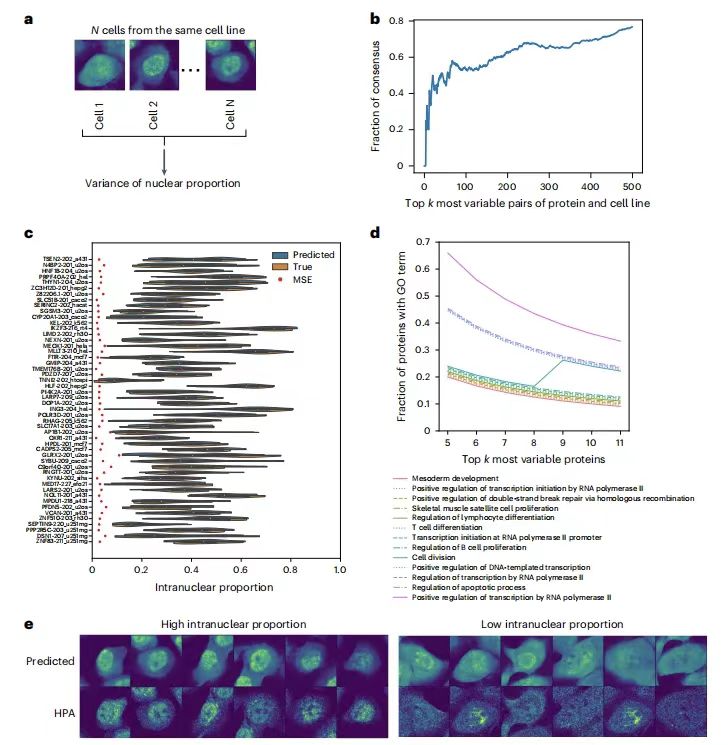
PUPS 可預測細胞系內單細胞中蛋白質定位的可變性
另外 Gene ontology(GO)分析表明,高度可變的蛋白質與細胞分裂、轉錄、雙鏈斷裂修復以及凋亡等過程有關。此外,
模型通過細胞標志性染色圖像捕捉形態(tài)等特征,表明了單細胞變異性不僅具有隨機性,還與細胞形態(tài)特征相關,為解釋單細胞異質性機制提供了新視角。
PUPS 在訓練數(shù)據(jù)之外的新實驗中的驗證
為了驗證 PUPS 在新的實驗環(huán)境下預測蛋白質定位的泛化能力,研究團隊選擇了 9 種蛋白質在 5 個細胞系中進行驗證。如下圖所示:

PUPS 在 HPA 之外的實驗中預測蛋白質亞細胞定位的能力
ATP13A5、CHID1、COPA、MESD 和 RBM23 為細胞系間變異最大的蛋白,它們都有不同的 GO term;DDIT3 和 N4BP2 是細胞系內單個細胞中變異最大的蛋白;EIF4G1 和 PSME3IP1 是細胞系間變異最小的蛋白,前者預計主要位于細胞核外,后者預計主要位于細胞核內。5 個細胞系中,除 A375 外,其他 HeLa、MCF7、GAMG 和 HEK293FT 均包含在 HPA 中。
結果顯示,
PUPS 預測的蛋白質圖像在視覺上與實驗測量的圖像相似。利用預測蛋白圖像計算的每個單細胞的核蛋白比例與實驗測量圖像計算的比例密切相關,pearson 相關系數(shù)為 0.767。這表明,
PUPS 可以用于定量預測以前沒有實驗測量或在訓練圖譜中使用的蛋白質的定位。
PUPS 學習到有意義的蛋白質和細胞表征
實驗證明,PUPS 在未知蛋白質和細胞系中預測蛋白質定位的能力來自于學習到了蛋白質序列和細胞標志性圖像的有意義表示。
研究團隊繪制了對應于 12,614 個基因的 40,622 個蛋白質形態(tài)的蛋白質序列表示,具有相似定位的蛋白質往往具有相似的序列表示。為進一步證明模型能識別有意義的蛋白質序列模式以及預測定位,研究團隊使用 Positional Shapley 方法計算了特定蛋白質中每個氨基酸殘基對預測各細胞區(qū)室標簽預測的重要性,如成功解釋了 N4BP2 核定位的預測變異性,也與 CUE 結構域通泛素結合可能改變亞細胞定位的報道相符。

PUPS學習有意義的蛋白質和細胞表征
除此了識別有意義的蛋白質序列基序外,
研究團隊進一步表明了 PUPS 從細胞標志性染色中學習單細胞的有意義表征。其將從標志性染色中學習到的單細胞圖像表示可視化,發(fā)現(xiàn)即使細胞系標簽沒有輸入到模型當中,同一細胞系的單細胞也具有相似的圖像表示。蛋白質和細胞標志性圖像的聯(lián)合表示保留了細胞系和蛋白質之間的分離,而每個細胞系內的不同蛋白質在不同細胞系之間的順序相似。給定聯(lián)合表示空間中每個細胞系的質心,從質心到特定蛋白質的向量在所有細胞系中大部分是平行的,即在給定序列表示的情況下,預測特定蛋白質的圖像需要再表示空間中以相同方向移動,而不管細胞系是什么,
這解釋了 PUPS 通過學習有意義的蛋白質和細胞圖像表示來推廣到未知蛋白質和細胞系的能力。
此外,
PUPS 還能預測致病突變對蛋白質定位的影響。例如,針對核編碼的線粒體蛋白 SDHD 和 ETHE1 的突變研究表明,SDHD 突變會導致其核定位比例增加,這與疾病中核基因組不穩(wěn)定的機制一致;ETHE1 突變則顯示胞質定位比例升高,與已知的核 - 胞質穿梭異常相關。這些結果表明,PUPS 可通過分析序列變異對定位的影響,為疾病機制研究提供新線索。
蛋白質亞細胞定位預測新解
正如上述所言,蛋白質亞細胞定位預測在生物信息學和生物學研究中都具有重大意義,PUPS 提供了一種融合多模態(tài)信息的思路,為該領域的研究畫上了濃墨重彩的一筆。與此同時,該領域的研究經(jīng)過數(shù)十年的發(fā)展,其成果也早已是百花齊放。
愛爾蘭都柏林大學的團隊在 Computational and Structural Biotechology Journal 雜志上發(fā)表了一項研究,
其中圍繞蛋白質亞細胞定位預測介紹了多種計算方法,包括基于序列、注釋、混合及元預測等類別,同時文章還按真核生物、原核生物、病毒及多類別對亞細胞定位預測工具進行了分類介紹,真核生物預測工具如 mLASSO-Hum、DeepPSL 等,原核生物預測工具如 PRED-LIPO 等。通過設計涵蓋 7 個主要領域及 28 個子分類的機器學習和深度學習分類圖,該研究提供了單類別和多類別預測工具分類法,從而方便用戶查找方法、預測工具。論文以「Protein subcellular localization prediction tools」發(fā)表。
* 論文地址:
??https://www.sciencedirect.com/science/article/pii/S2001037024001156??
復旦大學生物醫(yī)學研究院楊力研究組與上海人工智能實驗室董楠卿研究組合作,于 4 月 12 日在 Briefings in Bioinformatics 雜志在線發(fā)表了題為「Deep Generative Model for Protein Subcellular Localization」的研究論文。
研究同樣基于 ESM2 蛋白質大語言模型及 U-Net 框架,開發(fā)了具備多模態(tài)處理能力的生成式深度學習模型 deepGPS。
據(jù)介紹,deepGPS 能夠接收蛋白質序列及細胞核圖像作為輸入,并生成蛋白質定位的文本標簽及分布圖像,是一種支持蛋白質亞細胞定位預測的新型「文生圖」(text-to-image)多模態(tài)模型。
* 論文地址:
隨著人工智能與生物學研究的融合加速,相關的創(chuàng)新性實驗也在不斷涌現(xiàn),并逐漸打破傳統(tǒng)方法的弊端,實現(xiàn)「兩全其美」甚至「十全十美」的表現(xiàn),從而推動生物信息學的快速發(fā)展。

















