Alphafold可能成為第一個獲諾貝爾獎的深度學習模型? 原創 精華
諾貝爾獎主要頒給那些為人類做出巨大貢獻的人,我國的屠呦呦就因發現青蒿素在2015年獲得諾貝爾醫學獎。
Alphafold不是某個人類科學家,它是Google DeepMind開發的一個深度學習模型,它能夠預測蛋白質結構。
把諾獎頒給一個模型,你可能會覺得我危言聳聽了,但如果你了解蛋白質結構的作用,你肯定會改變想法了。
一、蛋白質合成過程
在此之前還是先簡單了解下蛋白質的合成過程。
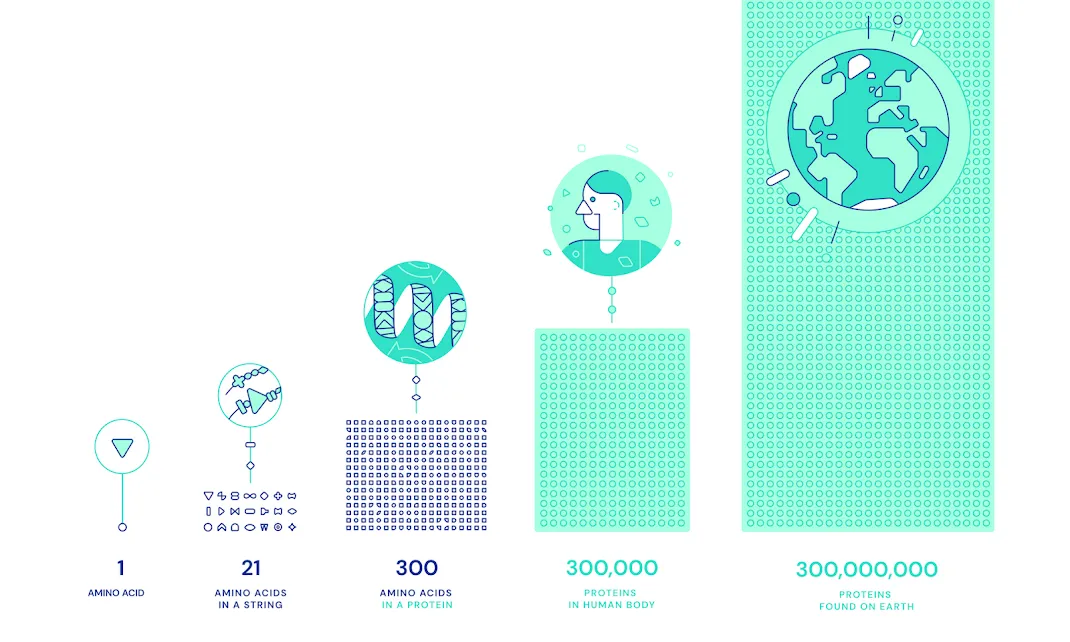
蛋白質是由氨基酸序列組成的,人體內有21種氨基酸種類,但這21種氨基酸能夠構成千萬種蛋白質,但目前已知結構的蛋白質才2萬個。
在人體細胞中,細胞核內有一個帶有遺傳信息的分子叫DNA,DNA中包含構造蛋白質的指令。
RNA通過一個被稱為轉錄的過程復制DNA信息,這個帶有遺傳信息的RNA被稱為mRNA,mRNA穿過細胞核來到細胞質內。
細胞質內還一個叫做核糖體的東西,蛋白質就是在這里合成的。
mRNA首先來到核糖體這里,細胞質內還有另一種RNA,叫做tRNA,tRNA一端是堿基對,另一端是氨基酸,當tRNA的堿基對和mRNA的堿基配對后,就會留下氨基酸,氨基酸就會串聯在一起,最后進行折疊,形成一個3D結構的蛋白質。
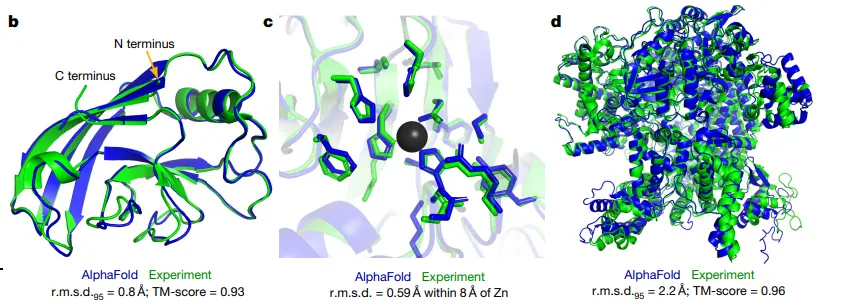
蛋白質合成是細胞分裂過程中的重要活動,細胞構成組織,組織構成器官。人體內的很多酶也是由蛋白質構成的,人體的眼睛為什么能感光?肝臟為什么能造血?這都與具有某種結構的蛋白質有關。
所以,了解了蛋白質的結構,也就知道了它的功能,進而進行疾病預防,藥物研發,甚至發現遺傳密碼。
二、傳統分析蛋白質折疊的方法
僅基于氨基酸序列預測蛋白質的3D結構難度是巨大的,約有10的143次方種折疊方式,這比宇宙中的原子個數還要多。
傳統預測蛋白質結構的方法主要有以下幾種:
- 同源建模(Homology modeling):也稱為比較建模或模板基礎建模,基于蛋白質的3D結構比它們的氨基酸序列更為保守的假設。這種方法主要使用序列比對和分子建模技術,通過已知結構的同源蛋白來預測目標蛋白的結構。
- 從頭建模(De novo modeling):這是一種基于“第一原理”的蛋白質結構預測方法,不依賴于已知的蛋白質結構,而是僅基于物理定律(量子力學)生成目標蛋白的3D結構。從頭建模方法通過設計的能量函數引導構象搜索,選擇具有最低能量的構象。
- 基于機器學習(ML)的建模:這是一種利用ML算法和已知蛋白質結構來預測目標蛋白質結構的策略。尤其是深度學習(DL)在這一領域取得了快速發展,DL基于大量數據和強大的計算能力,通過優化算法來預測蛋白質結構。
數百年來,為了預測蛋白質結構,需要借助磁共振核,X線等昂貴設備,同時需要消耗大量的時間進行實驗。
為了解決這個世紀難題,還成立了一個CASP挑戰賽。
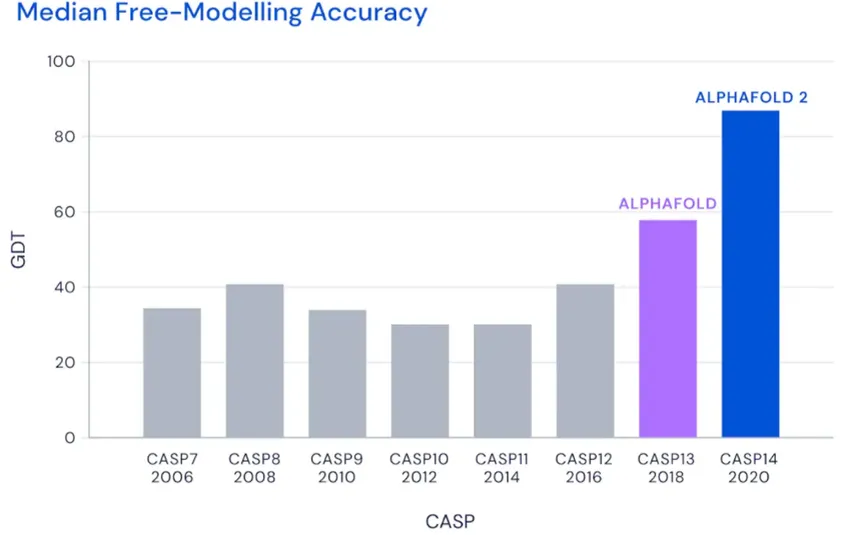
目前公認為,如果GDT達到85%就認為解決了蛋白質折疊世紀難題,如上圖所示,ALPHAFOLD2的效果已經接近這個目標。而它的前任也遠超傳統方法。
三、Alphafold2
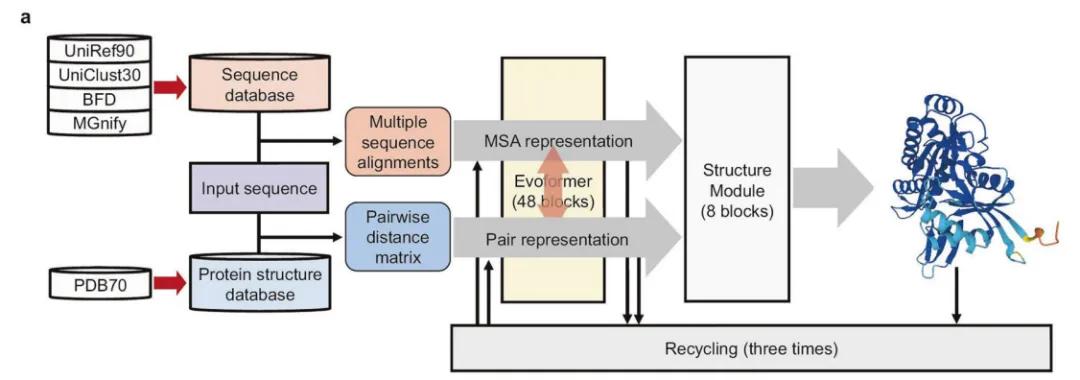
Alphafold2使用深度神經網絡利用同源蛋白和多序列比對的信息從氨基酸序列生成蛋白質結構。
Github:https://github.com/google-deepmind/alphafold
論文:https://www.nature.com/articles/s41586-021-03819-2
Colab Notebook:https://colab.research.google.com/github/sokrypton/ColabFold/blob/main/AlphaFold2.ipynb模型結構
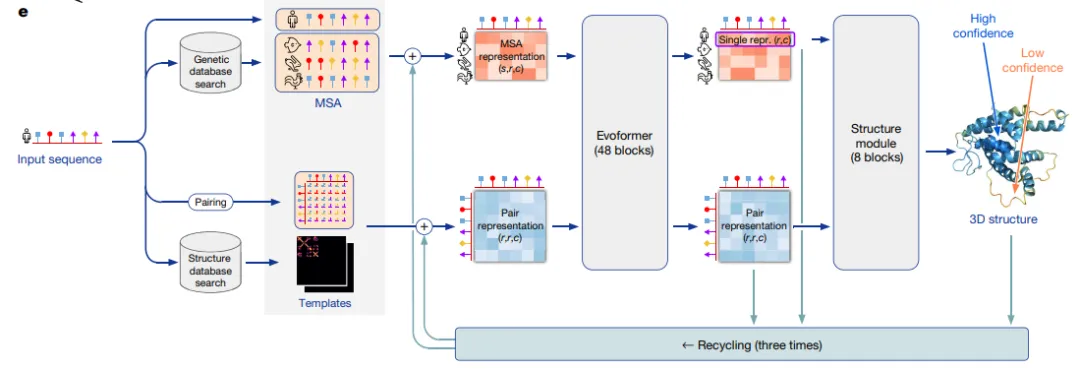
如下圖,首先根據輸入氨基酸序列和已知結構蛋白質數據庫生成MSA(多序列對比表示)和配對表示。
MSA表示和配對表示會被送入Evoformer,它實現了從輸入信息到蛋白質的3D原子坐標的轉換。
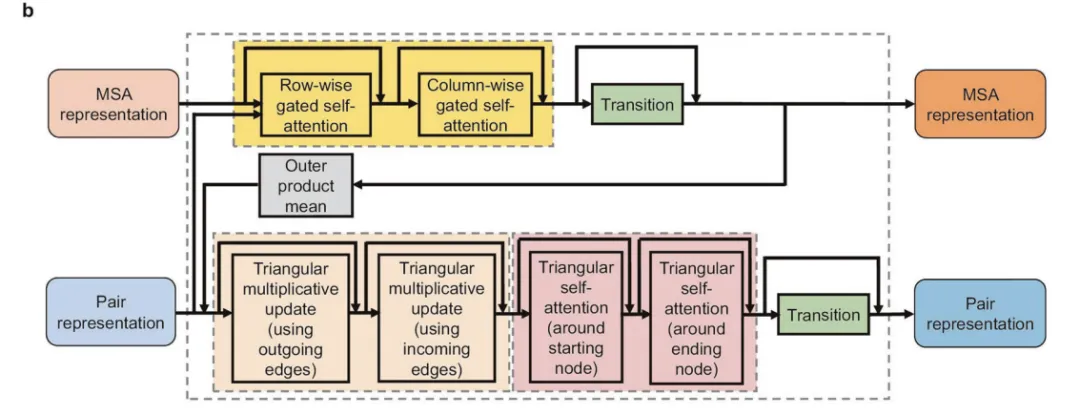
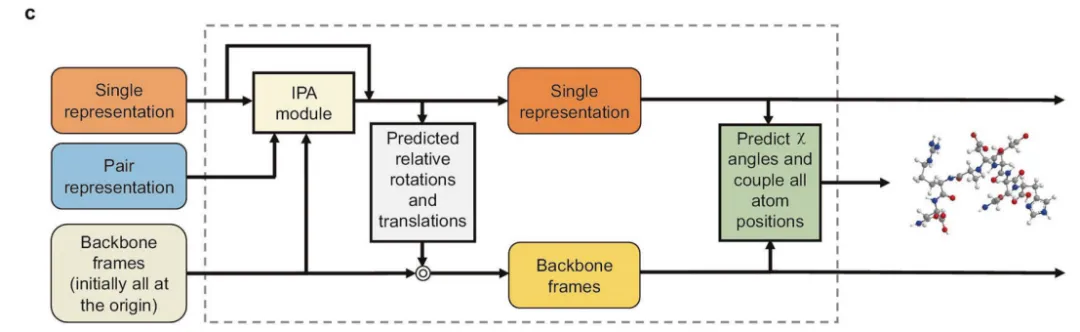
對于Alphafold2詳細信息,感興趣的可以去閱讀論文。
這里只是簡單概述一下,模型的輸入是組成蛋白質的氨基酸序列,輸出是PDB格式的數據,用于表示蛋白質3D結構信息。模型則是采用當下最流行的Transformer架構。
MSA是將輸入蛋白質氨基酸序列與已知結構的蛋白質進行對比,目的是找到與之相似的,然后基于這個相似的蛋白質結構來預測輸入蛋白質結構。
為了造福人類,DeepMind將一些蛋白質的預測結果收錄數據庫供其他組織或個人免費使用。
??https://alphafold.ebi.ac.uk??
本文轉載自公眾號人工智能大講堂

















