無限的可能性 NVIDIA生成式AI模型加速合成蛋白質
過去兩年,機器學習徹底改變了蛋白質結構預測。而現在,人工智能又在蛋白質設計領域引發了新一輪革命。
自從AI問世以來,眾多科學家們就加入了利用它來進行蛋白質研究的賽道。生物學家發現,使用機器學習,可以在幾秒鐘內創建出蛋白質分子。而在以前,這個時間也許是幾個月。
近日,初創企業Evozyne使用NVIDIA提供的預訓練AI模型,創造了兩種在醫療和清潔能源領域具有重大潛力的蛋白質。其中一種蛋白質用于治療一種先天性疾病,另一種用于消耗二氧化碳以減少全球變暖。
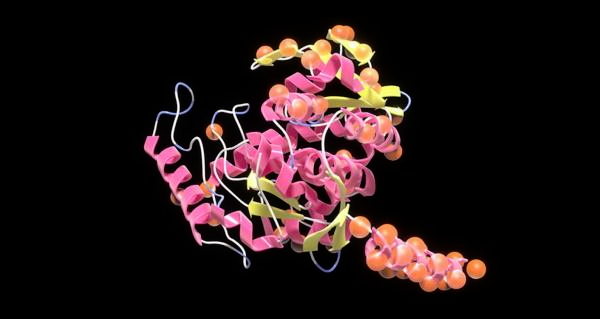
這樣科學家使用NVIDIA BioNeMo創建出能夠生成高質量蛋白質的大型語言模型,以此加快藥物研發并助力創造更具可持續性的環境。
加速藥物研發的新方法
Evozyne聯合創始人、論文共同作者Andrew Ferguson表示: “令人欣喜的是,這個AI模型第一輪產出的合成蛋白質就像自然生成的蛋白質一樣,表示該模型已經學會了自然界的設計規則。”
Evozyne使用了NVIDIA的ProtT5。ProtT5是一個Transformer模型,是用于創建醫療AI模型的軟件框架和服務——NVIDIA BioNeMo的一部分。
分子工程師Ferguson的研究領域涵蓋化學和機器學習領域,他表示:“BioNeMo非常強大,讓我們能夠訓練模型,然后以非常低的成本使用該模型來運行工作任務,在幾秒鐘內就能生成數百萬個序列。該模型預測了如何組裝出能夠滿足Evozyne需求的新蛋白質。”
該模型是Evovyne ProT-VAE流程的核心。Evozyne的ProT-VAE流程將NVIDIA BioNeMo中強大的Transformer模型與變分自編碼器(VAE)相結合。
他表示:“幾年前,還沒有人注意到可以使用大型語言模型與變分自編碼器相結合的方式來設計蛋白質。”
相比之下,Evozyne的方法只通過一輪,就能改變一個蛋白質中半數乃至以上的氨基酸。這相當于進行了數百次的突變。
Evozyne數據科學家Joshua Moller表示: “他們通過將工作擴展到多個GPU來加快訓練速度。
這將訓練大型 AI 模型的時間從幾個月縮短到一個星期。Ferguson表示:“所以我們能夠訓練出原本不可能訓練出的模型,比如一些有數十億可訓練參數的模型。”
革命性的AI模型
傳統的蛋白質工程設計方法,即定向進化,采用的是一種緩慢、無計劃的方法,通常一次只改變幾個氨基酸的序列。而機器學習有助于研究海量可能的氨基酸組合,然后有效地識別最有用的序列。
BioNeMo是一款基于NVIDIA NeMo Megatron構建的AI賦能藥物研發云服務和框架,用于在超級計算規模下訓練和部署大型生物分子Transformer AI模型。服務包括預訓練LLM、對蛋白質、DNA、RNA和化學的通用文件格式的原生支持,還提供可供SMILES(用于分子結構)和FASTA(用于氨基酸和核苷酸序列)使用的數據加載器。
借助BioNeMo,科學家可以開始為UniRef50和ZINC數據庫輕松使用預訓練模型、自動下載器和預處理器。借助無監督式結構化學習者,各種模型、嵌入和輸出得以結合,將多模態數據組合在一起。無監督式預訓練還消除了對已標記數據的需求,從而快速生成已學習的嵌入,預測蛋白質結構、功能、細胞位置、水溶性、膜結合性、保存區域和可變區域等。
其中,MegaMolBART是一種使用14億個分子(SMILES字符串)進行訓練的生成化學模型,可用于多種化學信息學應用。而且BioNeMo提供了ProtT5和ESM1-85M等基于 Transformer 的蛋白質語言模型。
BioNeMo還提供OpenFold,這是一種用于預測新型蛋白質序列 3D 結構的深度學習模型。
NVIDIA的Transformer模型會讀取數百萬種蛋白質中的氨基酸序列。該模型運用神經網絡用來理解文本的技術,學會了大自然如何構建蛋白質氨基酸序列。
展望未來,使用AI加速蛋白質工程的前景十分廣闊。人工設計出來的蛋白質相對于自然界中原本就存在的蛋白質而言,更加穩定,在沒有能量或高溫等極端情況下,也能實現它的某項功能。
此外,也可以用人工智能來設計氨基酸序列,使其與主干相符,用于改善酶、抗體等特定蛋白的穩定性。人工智能技術對于不同大小、不同構象的蛋白質設計起到了非常重要的作用,未來還可幫助設計更多、更有用的蛋白質,包括可用于減少污染、改善環境的新的生物材料。








































