斯坦福、微軟聯手,用擴散模型進行蛋白質結構生成,已開源
蛋白質對生命至關重要,幾乎在每個生物過程中都發揮著作用。一方面它們能在神經元之間傳遞信號、識別微觀入侵者并激活免疫反應等。另一方面,蛋白質作為一種治療介質已經得到廣泛研究,成為治療疾病的一部分。因此,通過生成新的、物理上可折疊的蛋白質結構,打開了利用細胞通路治療疾病新方法的大門。?
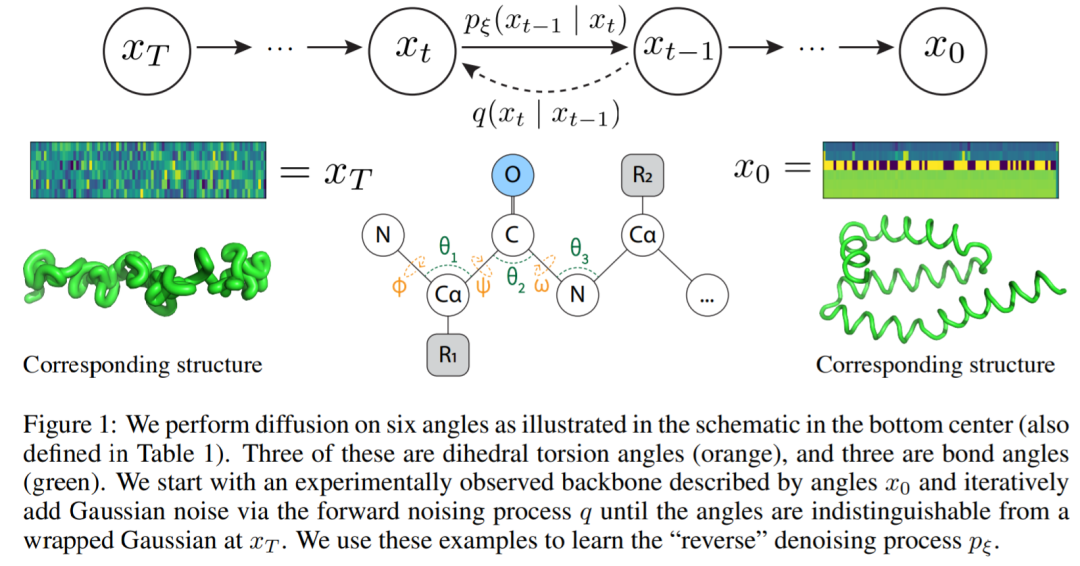
本文中,來自斯坦福大學、微軟研究院等機構的研究者,他們受體內蛋白質折疊過程的啟發,從而引入了一個折疊擴散( folding diffusion,FoldingDiff )模型,該模型通過鏡像蛋白質天然折疊過程來設計蛋白質主鏈結構。

- 論文地址:https://arxiv.org/pdf/2209.15611.pdf
- 代碼地址:https://github.com/microsoft/foldingdiff?
具體而言,他們將蛋白質主鏈結構描述為一系列連續的角度,以捕捉組成氨基酸殘基的相對方向,這種表示的固有位移和旋轉不變性極大地減輕了對復雜等變網絡的需要。
該研究基于 transformer 骨干訓練了一個去噪擴散概率模型,并證明本文的模型可以無條件地生成高度逼真的蛋白質結構,其復雜性和結構模式類似于天然蛋白質。
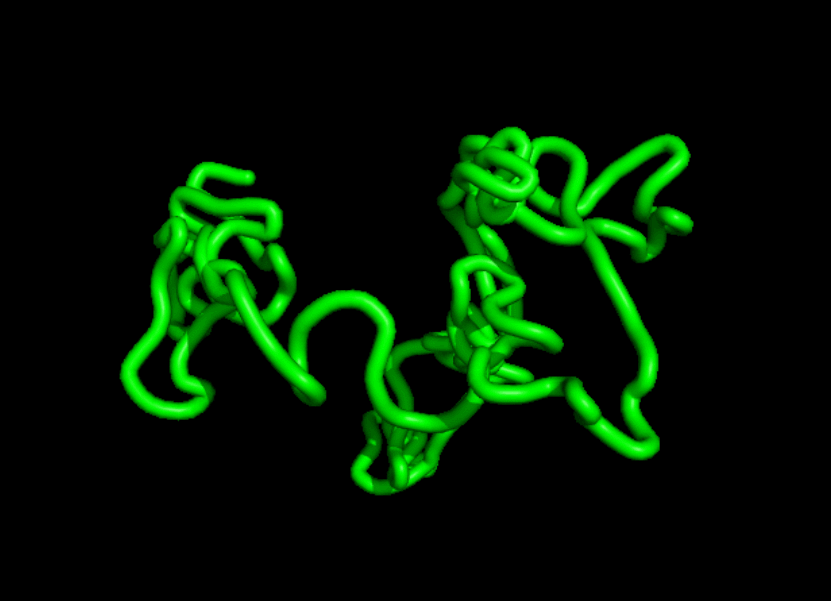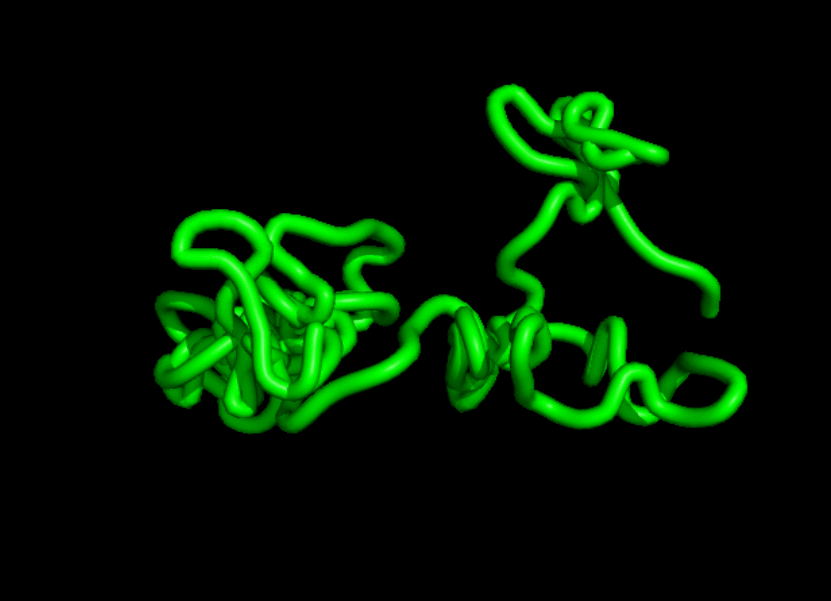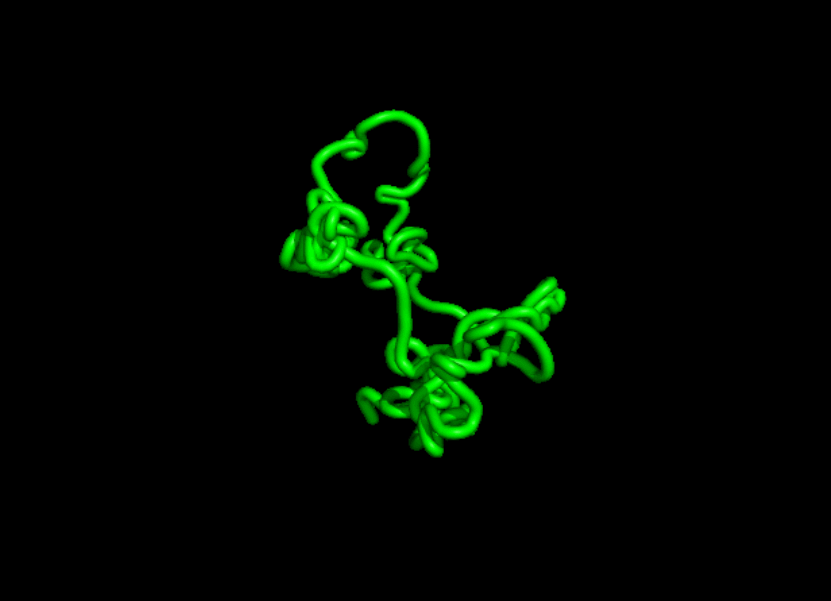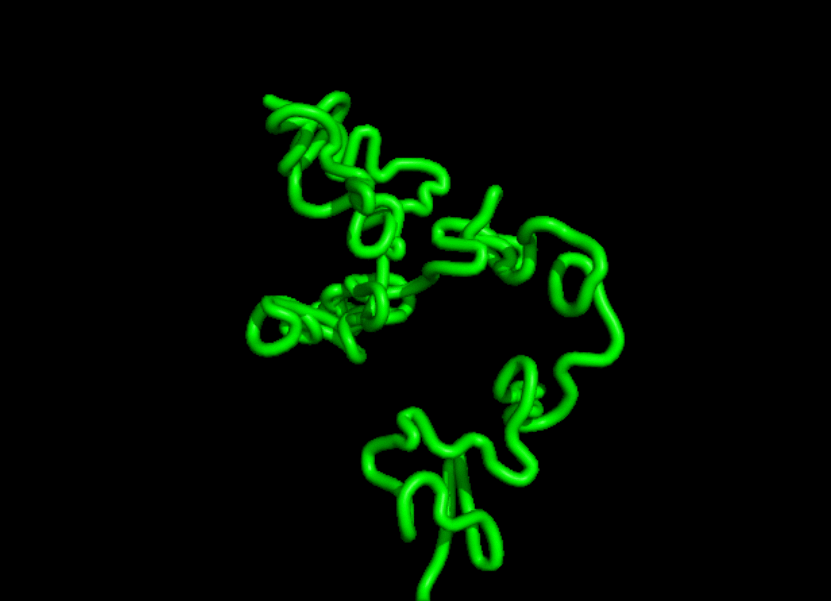
有網友表示:不知道這個模型是否會為 AlphaFold 帶來一些競爭。
方法及結果?
我們可以將蛋白質理解為是可變長度的氨基酸殘基鏈,典型氨基酸有 20 種,享有相同的三原子 N - C_α - C 主鏈,但具有不同的側鏈連接到 C_α 原子上(通常表示為 R,參見圖 1 所示 )。
這些殘基組裝形成聚合物鏈,折疊成 3D 結構,其形狀在很大程度上決定了蛋白質的功能。這些折疊結構可以用四個層次來描述:
- 一級結構,簡單地捕捉氨基酸的線性序列;
- 二級結構,描述氨基酸的局部排列;
- 三級結構,描述所有殘基的完整空間排列;
- 四級結構,描述多個不同的氨基酸鏈如何聚集在一起形成更大的復合物。
該研究提出了一個簡化的蛋白質主鏈框架,它遵循蛋白質折疊的生物學過程,同時消除了復雜的等變網絡的需要。與其將長度為 N 個氨基酸的蛋白質主鏈視為三維坐標,他們把它看作由六個內角、連續角組成的序列。也就是說,給定當前殘基的位置,六個內角的向量描述了下一個殘基中所有主鏈原子的相對位置。這些內角可以使用三角函數輕松計算,迭代地將原子添加到蛋白質骨架中,然后在轉換回 3D 笛卡爾坐標。
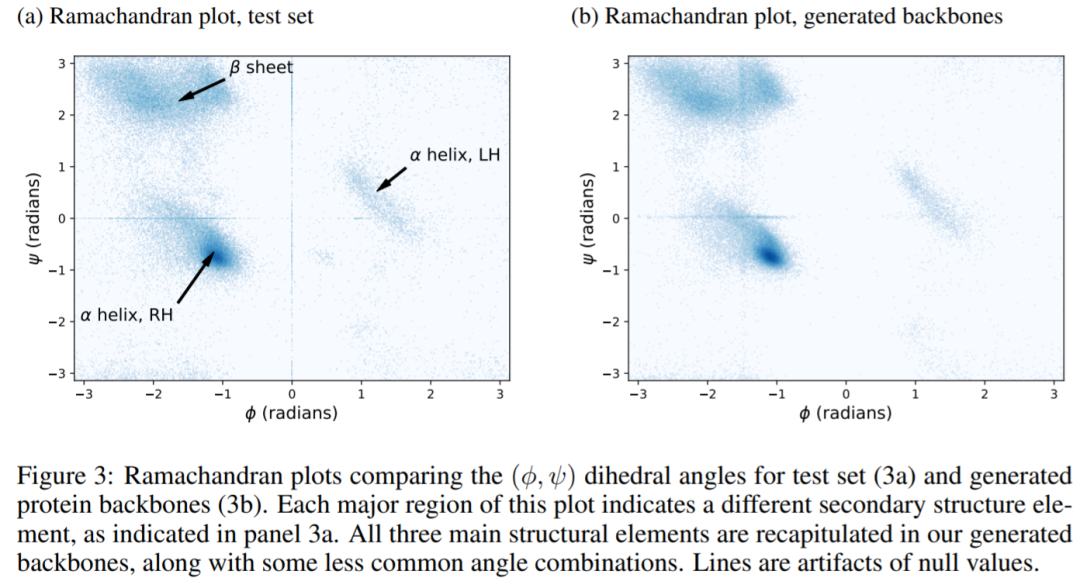
下圖為一項實驗結果。自然結構的 Ramachandran 圖(圖 a)包含三個區域,分別對應于 LH α螺旋、RH α 螺旋和β sheet。所有這三個區域都在本文生成的結構中全部再現(圖 3b)。換句話說,FoldingDiff 能夠生成蛋白質主鏈中的二級結構元素。此外,實驗還表明 FoldingDiff 模型能夠正確地了解到 RH α 螺旋比 LH α 螺旋更常見。而先前的工作使用等變網絡,是無法區分這兩種類型的螺旋的。
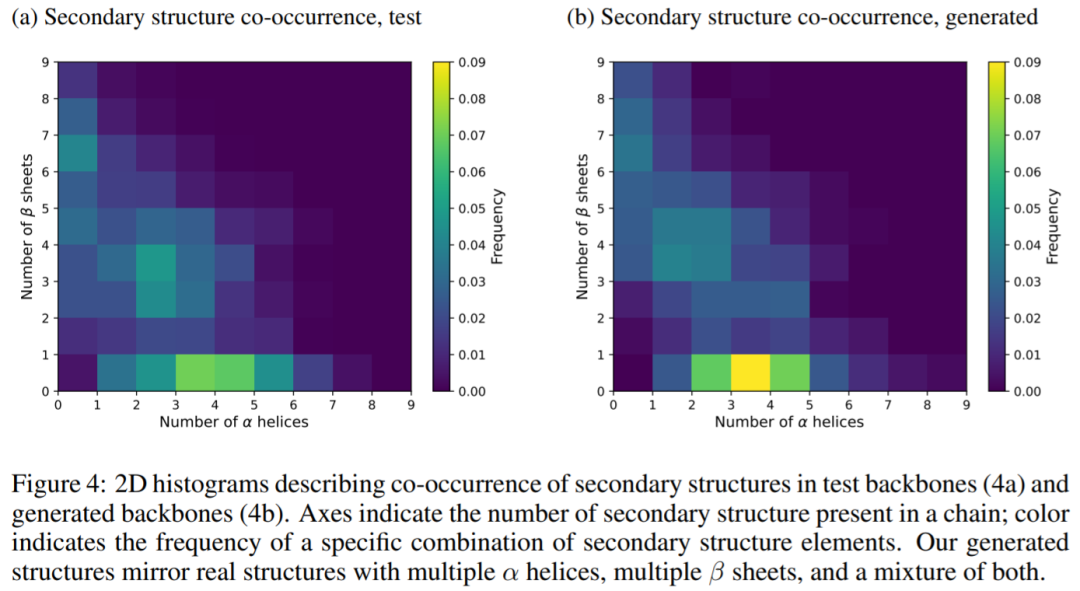
下圖為在測試主鏈 (4a) 和生成主鏈 (4b) 中出現二級結構的二維直方圖,結果表明生成的結構反映了蛋白質真實的結構,有多個α螺旋,多個β sheets ,以及兩者的混合。
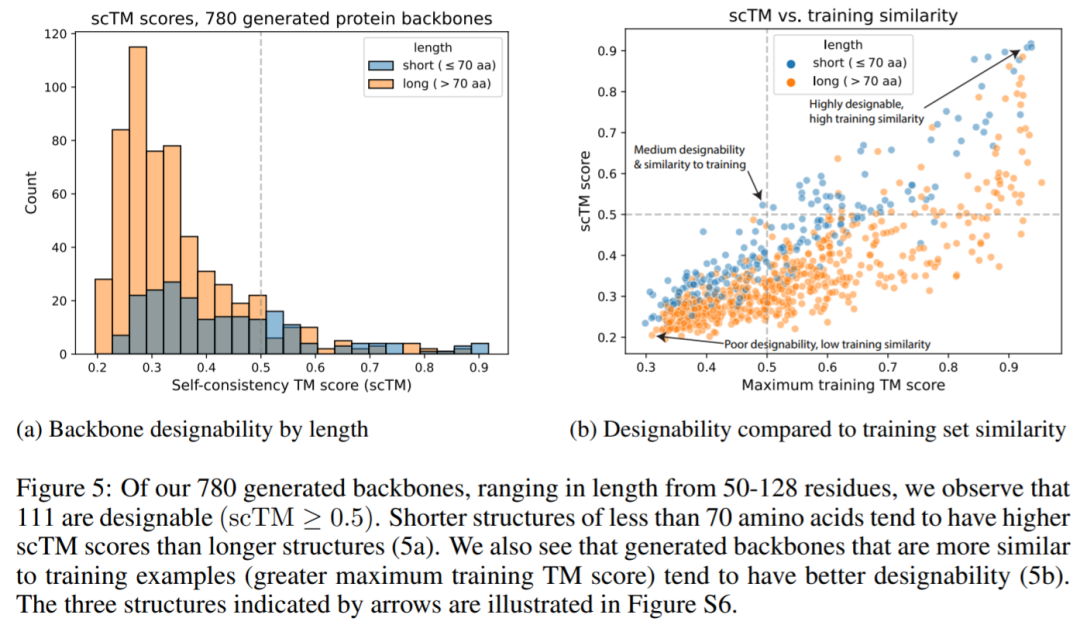
下圖表明,在生成的 780 個結構中有 111 個(占比為 14.2%)是可設計的,其 scTM 評分≥0.5(圖 5a),高于 Trippe 等人報告的 11.8% 的值。我們還看到,生成的主鏈與訓練示例更相似,往往具有更好的可設計性(5b)。
了解更多內容,請閱讀原論文。