利用進化擴散進行蛋白生成,微軟開源新型蛋白質生成AI框架EvoDiff
進化產生了多種功能蛋白,可以精確調節細胞過程。近年來出現了深度生成模型,旨在從這種多樣性中學習,生成既有效又新穎的蛋白質,最終目標是定制功能以解決當今突出的挑戰。
當涉及到在計算機上創造新的蛋白質時,深度生成模型正成為越來越強大的工具。擴散模型是一類生成模型,最近被證明可以生成生理上合理的蛋白質,與自然界中看到的任何實際蛋白質不同,它可以在從頭蛋白質設計中提供無與倫比的能力和控制。
然而,當前最先進的模型構建蛋白質結構,這嚴重限制了其訓練數據的廣度,并將代限制在蛋白質設計空間的微小且有偏差的部分。
微軟研究人員開發了 EvoDiff——一種通用擴散框架,通過將進化規模數據與擴散模型的獨特調節能力相結合,可以在序列空間中創建可調節的蛋白質。EvoDiff 可以使結構合理的蛋白質多樣化,涵蓋所有可能的序列和功能。EvoDiff 可以構建基于結構的模型無法訪問的蛋白質,例如那些具有無序部分的蛋白質,同時能夠為有用的結構基序設計支架,這一事實證明了基于序列的公式的普遍性。
在蛋白質序列進化中,EvoDiff 是第一個展示擴散生成模型功效的深度學習框架。
EvoDiff 的共同作者、微軟高級研究員 Ava Amini 表示:「如果有什么可以從 EvoDiff 中學到的,我認為那就是,我們可以而且應該通過序列進行蛋白質生成,因為我們能夠實現通用性、規模化和模塊化。我們的擴散框架使我們有能力做到這一點,并控制我們如何設計這些蛋白質以滿足特定的功能目標。」
EvoDiff 的另一位共同作者 Kevin K. Yang 表示,「我們設想 EvoDiff 將擴展蛋白質工程的能力,超越結構-功能范式,轉向可編程、序列優先的設計,通過 EvoDiff,我們證明我們實際上可能不需要結構,而是『蛋白質序列就是你所需要的』來可控地設計新蛋白質。」
該研究以《Protein generation with evolutionary diffusion: sequence is all you need》為題,發布在 bioRxiv 預印平臺上。

GitHub 地址:https://github.com/microsoft/evodiff
論文鏈接:https://doi.org/10.1101/2023.09.11.556673
6.4 億個參數
EvoDiff 框架的核心是一個包含 6.4 億個參數的模型,該模型根據來自所有不同物種和蛋白質功能類別的數據進行訓練。訓練模型的數據來源于用于序列比對的 OpenFold 數據集 UniRef50,UniProt 數據的子集,UniProt 聯盟維護的蛋白質序列和功能信息數據庫。
Uniref50 是一個包含約 4200 萬個蛋白質序列的數據集。MSA 來自 OpenFold 數據集,其中包括 16,000,000 個 UniClust30 集群和 401,381 個 MSA,涵蓋 140,000 個不同的 PDB 鏈。有關 IDR 的信息來自反向同源 GitHub。
EvoDiff 的主要特征
EvoDiff 主要特征如下:
- 為了生成可管理的蛋白質序列,EvoDiff 將進化規模數據與擴散模型相結合。
- EvoDiff 可以使結構合理的蛋白質多樣化,涵蓋所有可能的序列和功能。
- 除了生成具有無序部分和基于結構的模型無法獲得的其他特征的蛋白質外,EvoDiff 還可以生成功能性結構基序的支架,證明了基于序列的配方的普遍適用性。
EvoDiff 是一種新穎的生成建模系統,用于僅從序列數據創建可編程蛋白質,該系統是通過將進化規模數據集與擴散模型相結合而開發的。其使用離散擴散框架,其中正向過程通過改變其氨基酸特性來迭代地破壞蛋白質序列,并且由神經網絡參數化的學習反向過程利用自然框架來預測每次迭代時所做的變化。蛋白質作為氨基酸語言上離散標記的序列。
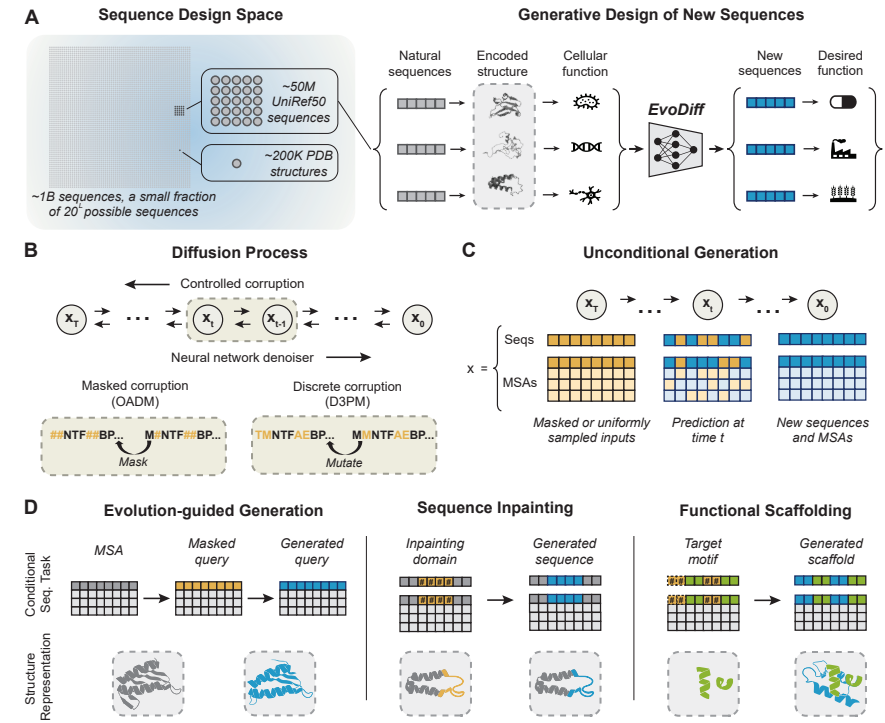
圖 1:EvoDiff,用于僅從序列數據進行可控蛋白質設計。(來源:論文)
可以使用反向方法從頭開始創建蛋白質序列。與蛋白質結構設計中傳統使用的連續擴散公式相比,EvoDiff 中使用的離散擴散公式在數學上取得了顯著的改進。多重序列比對 (MSA) 突出顯示相關蛋白質組的氨基酸序列的保守模式和變異,從而捕獲單個蛋白質序列進化規模數據集之外的進化聯系。為了利用這種額外深度的進化信息,他們構建了在 MSA 上訓練的離散擴散模型,以產生新穎的 single lines。
在序列空間中創建可調節的蛋白質
為了說明其在可調蛋白質設計方面的功效,研究人員在一系列生成活動的譜上檢查了序列和 MSA 模型(分別為 EvoDiff-Seq 和 EvoDiff-MSA)。
他們首先證明 EvoDiff-Seq 能夠可靠地產生高質量、多樣化的蛋白質,準確反映自然界蛋白質的組成和功能。EvoDiff-MSA 通過比對具有相似但獨特進化歷史的蛋白質來引導新序列的開發。最后,他們表明 EvoDiff 可以可靠地生成具有 IDR 的蛋白質,直接克服基于結構的生成模型的關鍵限制,并且可以通過利用基于擴散的建模框架的調節功能,在沒有任何明確結構信息的情況下生成功能結構基序的支架。
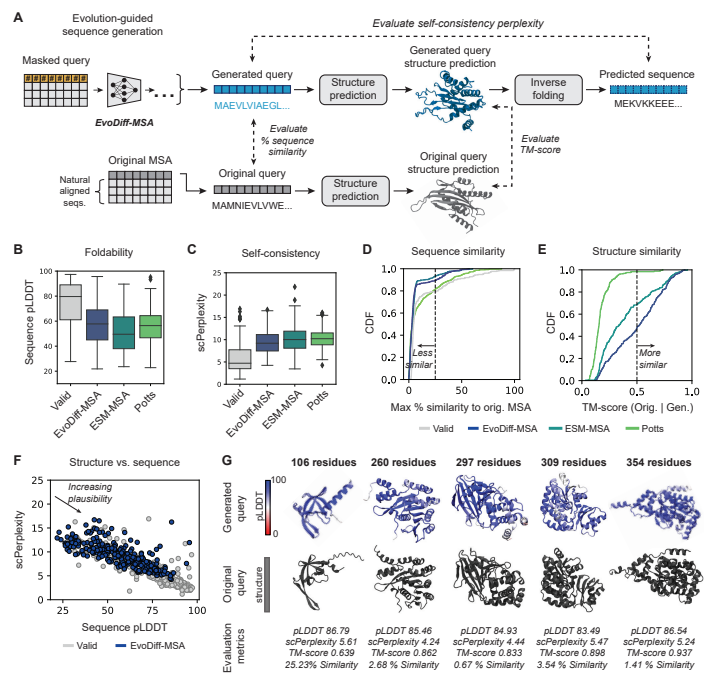
圖 2:EvoDiff-MSA 支持進化引導序列生成。(來源:論文)
為了生成具有基于序列限制條件調節的可能性的多樣化新蛋白質,研究人員提出了 EvoDiff,一種擴散建模框架。通過挑戰基于結構的蛋白質設計范式,EvoDiff 可以通過從序列數據生成本質上無序的區域和支架結構基序,無條件地采樣結構合理的蛋白質多樣性。
通過引導進行調節,創建的序列可以迭代地調整以滿足所需的質量,可以在未來的研究中添加到這些功能中。EvoDiff-D3PM 框架很適合通過指導進行條件調節,因為序列中每個殘基的身份都可以在每個解碼步驟中進行編輯。
然而,研究人員觀察到,OADM 在無條件生成方面通常優于 D3PM,這可能是因為 OADM 去噪任務比 D3PM 更容易學習。不幸的是,OADM 和其他現有的條件 LRAR 模型(如 ProGen)降低了指導的有效性。預計新的蛋白質序列將通過調節 EvoDiff-D3PM 的功能目標(例如序列功能分類器描述的目標)來生成。
EvoDiff 數據要求極低
EvoDiff 的數據要求極低,這意味著它可以輕松適應后續用途,而這只有通過基于結構的方法才能實現。研究人員表明,EvoDiff 可以通過修復來創建 IDR,無需微調,從而避免了基于結構的預測和生成模型的經典陷阱。
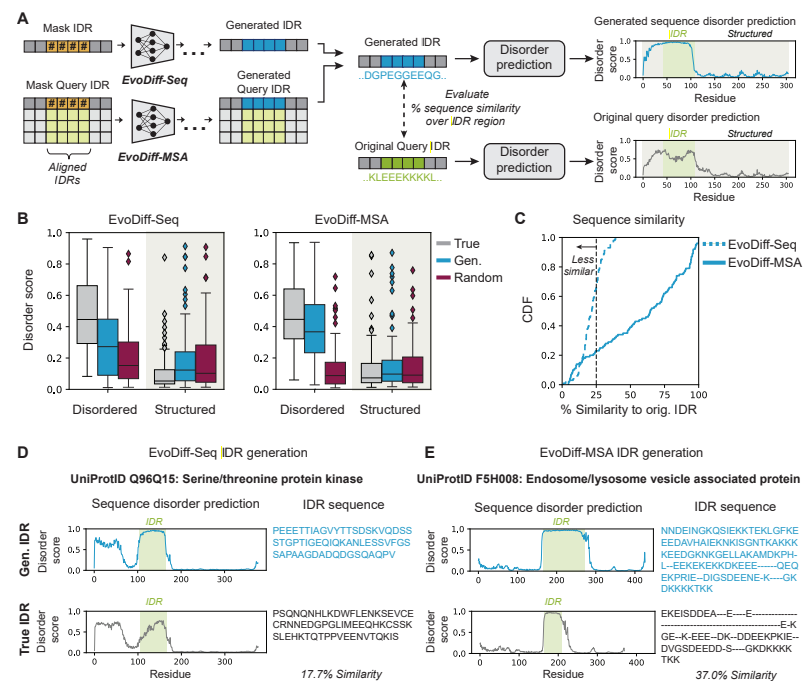
圖 3:EvoDiff 生成本質上無序的區域。(來源:論文)
獲取大型測序數據集結構的高昂成本可能會阻止研究人員使用新的生物、醫學或科學設計選項,這些選項可以通過在特定于應用程序的數據集(例如來自顯示庫或大型屏幕的數據集)上微調 EvoDiff 來解鎖。盡管 AlphaFold 和相關算法可以預測許多序列的結構,但它們在點突變方面遇到困難,并且在指示虛假蛋白質的結構時可能過于自信。
下一步計劃
總之,微軟科學家發布了一套離散擴散模型,可用于進行基于序列的蛋白質工程和設計。可以擴展 EvoDiff 模型以進行基于結構或功能的引導設計,并且它們可以立即用于無條件、進化引導和條件創建蛋白質序列。他們希望通過直接用蛋白質語言讀取和寫入過程,EvoDiff 將為可編程蛋白質創造開辟新的可能性。
「這只是一個包含 6.4 億參數的模型,如果我們擴展到數十億個參數,我們可能會看到生成質量的提高,」Alamdari 說道。「雖然我們演示了一些粗粒度的策略,但為了實現更細粒度的控制,我們希望根據文本、化學信息或其他方式來調節 EvoDiff 來指定所需的功能。」
下一步,EvoDiff 團隊計劃測試模型在實驗室生成的蛋白質,以確定它們是否可行。如果事實證明是這樣,他們將開始開發下一代框架。