醫學圖像的深度學習的完整代碼示例:使用Pytorch對MRI腦掃描的圖像進行分割
圖像分割是醫學圖像分析中最重要的任務之一,在許多臨床應用中往往是第一步也是最關鍵的一步。在腦MRI分析中,圖像分割通常用于測量和可視化解剖結構,分析大腦變化,描繪病理區域以及手術計劃和圖像引導干預,分割是大多數形態學分析的先決條件。
本文我們將介紹如何使用QuickNAT對人腦的圖像進行分割。使用MONAI, PyTorch和用于數據可視化和計算的常見Python庫,如NumPy, TorchIO和matplotlib。
本文將主要設計以下幾個方面:
- 設置數據集和探索數據
- 處理和準備數據集適當的模型訓練
- 創建一個訓練循環
- 評估模型并分析結果
完整的代碼會在本文最后提供。
設置數據目錄
使用MONAI的第一步是設置MONAI_DATA_DIRECTORY環境變量指定目錄,如果未指定將使用臨時目錄。
directory = os.environ.get("MONAI_DATA_DIRECTORY")
root_dir = tempfile.mkdtemp() if directory is None else directory
print(root_dir)設置數據集
將CNN模型擴展到大腦分割的主要挑戰之一是人工注釋的訓練數據的有限性。作者引入了一種新的訓練策略,利用沒有手動標簽的大型數據集和有手動標簽的小型數據集。
首先,使用現有的軟件工具(例如FreeSurfer)從大型未標記數據集中獲得自動生成的分割,然后使用這些工具對網絡進行預訓練。在第二步中,使用更小的手動注釋數據[2]對網絡進行微調。
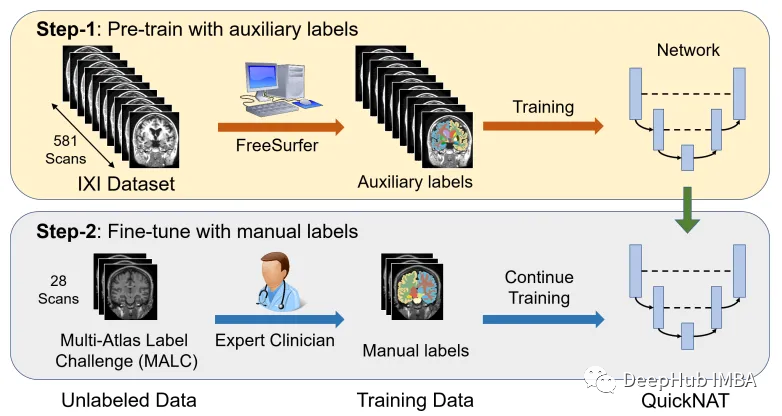
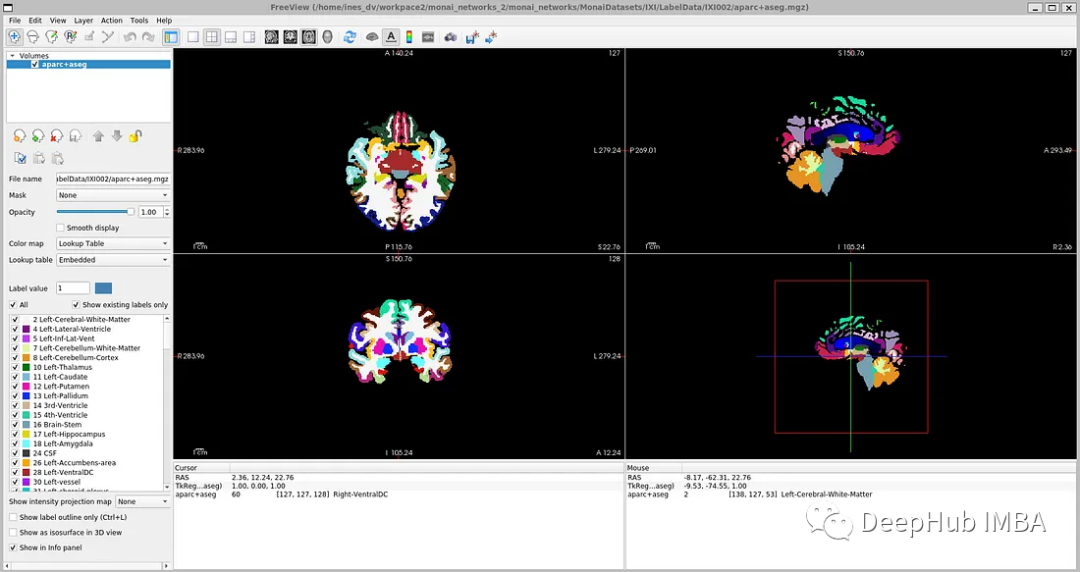
IXI數據集由581個健康受試者的未標記MRI T1掃描組成。這些數據是從倫敦3家不同的醫院收集來的。使用該數據集的主要缺點是標簽不是公開可用的,因此為了遵循與研究論文中相同的方法,本文將使用FreeSurfer為這些MRI T1掃描生成分割。
FreeSurfer是一個用于分析和可視化結構的軟件包。下載和安裝說明可以在這里找到。可以直接使用了“recon-all”命令來執行所有皮層重建過程。
盡管FreeSurfer是一個非常有用的工具,可以利用大量未標記的數據,并以監督的方式訓練網絡,但是掃描生成這些標簽需要長達5個小時,所以我們這里直接使用OASIS數據集來訓練模型,OASIS數據集是一個較小的數據集,具有公開可用的手動注釋。
OASIS是一個向科學界免費提供大腦神經成像數據集的項目。OASIS-1是由39個受試者的橫斷面組成的數據集,獲取方式如下:
resource = "https://download.nrg.wustl.edu/data/oasis_cross-sectional_disc1.tar.gz"
md5 = "c83e216ef8654a7cc9e2a30a4cdbe0cc"
compressed_file = os.path.join(root_dir, "oasis_cross-sectional_disc1.tar.gz")
data_dir = os.path.join(root_dir, "Oasis_Data")
if not os.path.exists(data_dir):
download_and_extract(resource, compressed_file, data_dir, md5)數據探索
如果你打開' oasis_crosssectional_disc1 .tar.gz ',你會發現每個主題都有不同的文件夾。例如,對于主題OAS1_0001_MR1,是這樣的:
鏡像數據文件路徑:disc1\OAS1_0001_MR1\PROCESSED\MPRAGE\T88_111\ oas1_0001_mr1_mpr_n4_anon_111_t88_masked_ggc .img
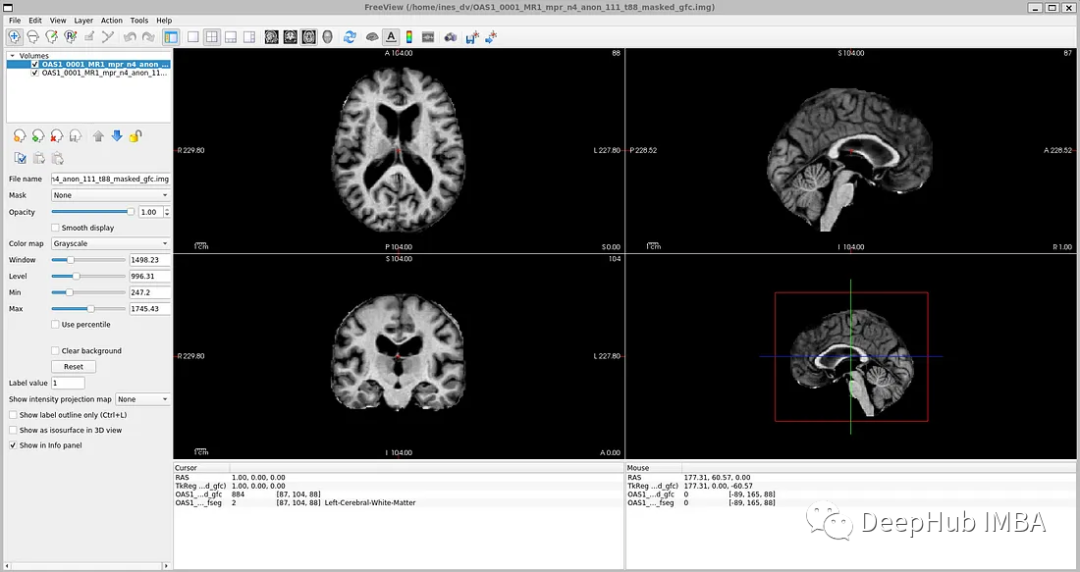
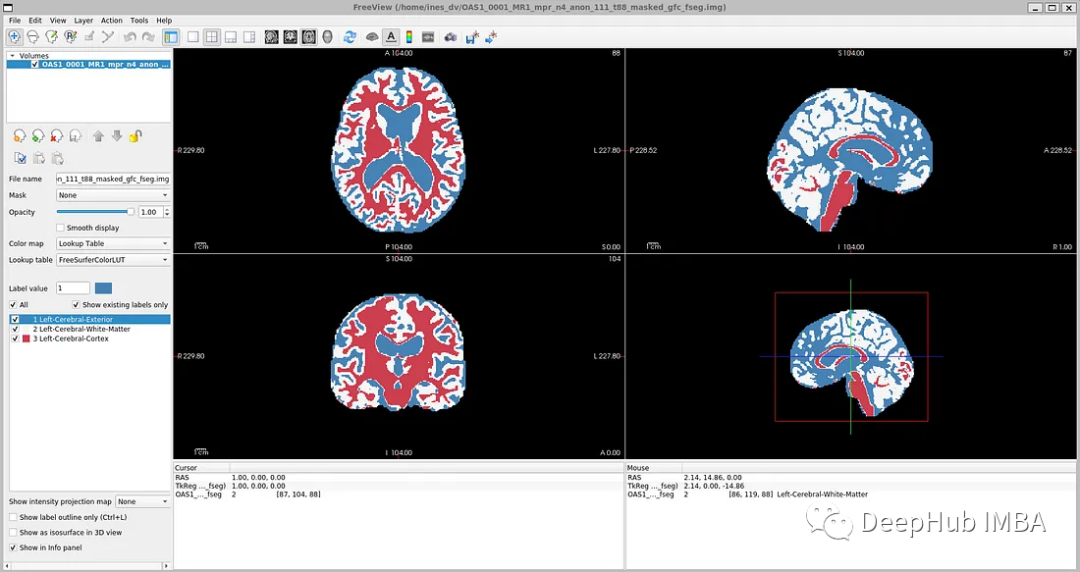
標簽文件:disc1\OAS1_0001_MR1\FSL_SEG\OAS1_0001_MR1_mpr_n4_anon_111_t88_masked_gfc_fseg.img
數據加載和預處理
下載數據集并將其提取到臨時目錄后,需要對其進行重構,我們希望我們的目錄看起來像這樣:
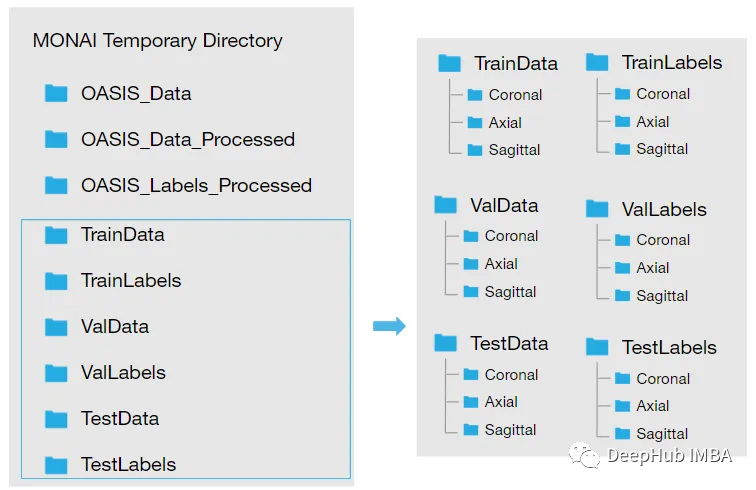
所以需要按照下面的步驟加載數據:
將。img文件轉換為。nii文件并保存到新文件夾中:創建兩個新文件夾。Oasis_Data_Processed包括每個受試者的處理過的MRI T1掃描,Oasis_Labels_Processed包括相應的標簽。
new_path_data= root_dir + '/Oasis_Data_Processed/'
if not os.path.exists(new_path_data):
os.makedirs(new_path_data)
new_path_labels= root_dir + '/Oasis_Labels_Processed/'
if not os.path.exists(new_path_labels):
os.makedirs(new_path_labels)然后就是對其進行操作:
for i in [x for x in range(1, 43) if x != 8 and x != 24 and x != 36]:
if i < 7 or i == 9:
filename = root_dir + '/Oasis_Data/disc1/OAS1_000'+ str(i) + '_MR1/PROCESSED/MPRAGE/T88_111/OAS1_000' + str(i) + '_MR1_mpr_n4_anon_111_t88_masked_gfc.img'
elif i == 7:
filename = root_dir + '/Oasis_Data/disc1/OAS1_000'+ str(i) + '_MR1/PROCESSED/MPRAGE/T88_111/OAS1_000' + str(i) + '_MR1_mpr_n3_anon_111_t88_masked_gfc.img'
elif i==15 or i==16 or i==20 or i==24 or i==26 or i==34 or i==38 or i==39:
filename = root_dir + '/Oasis_Data/disc1/OAS1_00'+ str(i) + '_MR1/PROCESSED/MPRAGE/T88_111/OAS1_00' + str(i) + '_MR1_mpr_n3_anon_111_t88_masked_gfc.img'
else:
filename = root_dir + '/Oasis_Data/disc1/OAS1_00'+ str(i) + '_MR1/PROCESSED/MPRAGE/T88_111/OAS1_00' + str(i) + '_MR1_mpr_n4_anon_111_t88_masked_gfc.img'
img = nib.load(filename)
nib.save(img, filename.replace('.img', '.nii'))
i = i+1具體代碼就不再粘貼了,有興趣的看看最后的完整代碼。下一步就是讀取圖像和標簽文件名
image_files = sorted(glob(os.path.join(root_dir + '/Oasis_Data_Processed', '*.nii')))
label_files = sorted(glob(os.path.join(root_dir + '/Oasis_Labels_Processed', '*.nii')))
files = [{'image': image_name, 'label': label_name} for image_name, label_name in zip(image_files, label_files)]為了可視化帶有相應標簽的圖像,可以使用TorchIO,這是一個Python庫,用于深度學習中多維醫學圖像的加載、預處理、增強和采樣。
image_filename = root_dir + '/Oasis_Data_Processed/OAS1_0001_MR1_mpr_n4_anon_111_t88_masked_gfc.nii'
label_filename = root_dir + '/Oasis_Labels_Processed/OAS1_0001_MR1_mpr_n4_anon_111_t88_masked_gfc_fseg.nii'
subject = torchio.Subject(image=torchio.ScalarImage(image_filename), label=torchio.LabelMap(label_filename))
subject.plot()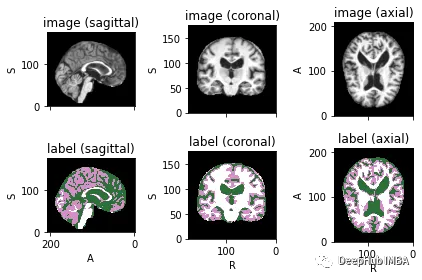
下面就是將數據分成3部分——訓練、驗證和測試。將數據分成三個不同的類別的目的是建立一個可靠的機器學習模型,避免過擬合。
我們將整個數據集分成三個部分:
Train: 80%,Validation: 10%,Test: 10%
train_inds, val_inds, test_inds = partition_dataset(data = np.arange(len(files)), ratios = [8, 1, 1], shuffle = True)
train = [files[i] for i in sorted(train_inds)]
val = [files[i] for i in sorted(val_inds)]
test = [files[i] for i in sorted(test_inds)]
print(f"Training count: {len(train)}, Validation count: {len(val)}, Test count: {len(test)}")因為模型需要的是二維切片,所以將每個切片保存在不同的文件夾中,如下圖所示。這兩個代碼單元將訓練集的每個MRI體積的切片保存為“.png”格式。
Save coronal slices for training images
dir = root_dir + '/TrainData'
os.makedirs(os.path.join(dir, "Coronal"))
path = root_dir + '/TrainData/Coronal/'
for file in sorted(glob(os.path.join(root_dir + '/TrainData', '*.nii'))):
image=torchio.ScalarImage(file)
data = image.data
filename = os.path.basename(file)
filename = os.path.splitext(filename)
for i in range(0, 208):
slice = data[0, :, i]
array = slice.numpy()
data_dir = root_dir + '/TrainData/Coronal/' + filename[0] + '_slice' + str(i) + '.png'
plt.imsave(fname = data_dir, arr = array, format = 'png', cmap = plt.cm.gray)同理,下面是保存標簽:
dir = root_dir + '/TrainLabels'
os.makedirs(os.path.join(dir, "Coronal"))
path = root_dir + '/TrainLabels/Coronal/'
for file in sorted(glob(os.path.join(root_dir + '/TrainLabels', '*.nii'))):
label = torchio.LabelMap(file)
data = label.data
filename = os.path.basename(file)
filename = os.path.splitext(filename)
for i in range(0, 208):
slice = data[0, :, i]
array = slice.numpy()
data_dir = root_dir + '/TrainLabels/Coronal/' + filename[0] + '_slice' + str(i) + '.png'
plt.imsave(fname = data_dir, arr = array, format = 'png')為訓練和驗證定義圖像的變換處理
在本例中,我們將使用Dictionary Transforms,其中數據是Python字典。
train_images_coronal = []
for file in sorted(glob(os.path.join(root_dir + '/TrainData/Coronal', '*.png'))):
train_images_coronal.append(file)
train_images_coronal = natsort.natsorted(train_images_coronal)
train_labels_coronal = []
for file in sorted(glob(os.path.join(root_dir + '/TrainLabels/Coronal', '*.png'))):
train_labels_coronal.append(file)
train_labels_coronal= natsort.natsorted(train_labels_coronal)
val_images_coronal = []
for file in sorted(glob(os.path.join(root_dir + '/ValData/Coronal', '*.png'))):
val_images_coronal.append(file)
val_images_coronal = natsort.natsorted(val_images_coronal)
val_labels_coronal = []
for file in sorted(glob(os.path.join(root_dir + '/ValLabels/Coronal', '*.png'))):
val_labels_coronal.append(file)
val_labels_coronal = natsort.natsorted(val_labels_coronal)
train_files_coronal = [{'image': image_name, 'label': label_name} for image_name, label_name in zip(train_images_coronal, train_labels_coronal)]
val_files_coronal = [{'image': image_name, 'label': label_name} for image_name, label_name in zip(val_images_coronal, val_labels_coronal)]現在我們將應用以下變換:
LoadImaged:加載圖像數據和元數據。我們使用' PILReader '來加載圖像和標簽文件。ensure_channel_first設置為True,將圖像數組形狀轉換為通道優先。
Rotate90d:我們將圖像和標簽旋轉90度,因為當我們下載它們時,它們方向是不正確的。
ToTensord:將輸入的圖像和標簽轉換為張量。
NormalizeIntensityd:對輸入進行規范化。
train_transforms = Compose(
[
LoadImaged(keys = ['image', 'label'], reader=PILReader(converter=lambda image: image.convert("L")), ensure_channel_first = True),
Rotate90d(keys = ['image', 'label'], k = 2),
ToTensord(keys = ['image', 'label']),
NormalizeIntensityd(keys = ['image'])
]
)
val_transforms = Compose(
[
LoadImaged(keys = ['image', 'label'], reader=PILReader(converter=lambda image: image.convert("L")), ensure_channel_first = True),
Rotate90d(keys = ['image', 'label'], k = 2),
ToTensord(keys = ['image', 'label']),
NormalizeIntensityd(keys = ['image'])
]
)MaskColorMap將我們定義了一個新的轉換,將相應的像素值以一種格式映射為多個標簽。這種轉換在語義分割中是必不可少的,因為我們必須為每個可能的類別提供二元特征。One-Hot Encoding將對應于原始類別的每個樣本的特征賦值為1。
因為OASIS-1數據集只有3個大腦結構標簽,對于更詳細的分割,理想的情況是像他們在研究論文中那樣對28個皮質結構進行注釋。在OASIS-1下載說明中,可以找到使用FreeSurfer獲得的更多大腦結構的標簽。
所以本文將分割更多的神經解剖結構。我們要將模型的參數num_classes修改為相應的標簽數量,以便模型的輸出是具有N個通道的特征映射,等于num_classes。
為了簡化本教程,我們將使用以下標簽,比OASIS-1但是要比FreeSurfer的少:
- Label 0: Background
- Label 1: LeftCerebralExterior
- Label 2: LeftWhiteMatter
- Label 3: LeftCerebralCortex
所以MaskColorMap的代碼如下:
class MaskColorMap(Enum):
Background = (30)
LeftCerebralExterior = (91)
LeftWhiteMatter = (137)
LeftCerebralCortex = (215)數據集和數據加載
數據集和數據加載器從存儲中提取數據,并將其分批發送給訓練循環。這里我們使用monai.data.Dataset加載之前定義的訓練和驗證字典,并對輸入數據應用相應的轉換。dataloader用于將數據集加載到內存中。我們將為訓練和驗證以及每個視圖定義一個數據集和數據加載器。
為了方便演示,我們使用通過使用torch.utils.data.Subset,在指定的索引處創建一個子集,只是用部分數據訓練加快演示速度。
train_dataset_coronal = Dataset(data=train_files_coronal, transform = train_transforms)
train_loader_coronal = DataLoader(train_dataset_coronal, batch_size = 1, shuffle = True)
val_dataset_coronal = Dataset(data = val_files_coronal, transform = val_transforms)
val_loader_coronal = DataLoader(val_dataset_coronal, batch_size = 1, shuffle = False)
# We will use a subset of the dataset
subset_train = list(range(90, len(train_dataset_coronal), 120))
train_dataset_coronal_subset = torch.utils.data.Subset(train_dataset_coronal, subset_train)
train_loader_coronal_subset = DataLoader(train_dataset_coronal_subset, batch_size = 1, shuffle = True)
subset_val = list(range(90, len(val_dataset_coronal), 50))
val_dataset_coronal_subset = torch.utils.data.Subset(val_dataset_coronal, subset_val)
val_loader_coronal_subset = DataLoader(val_dataset_coronal_subset, batch_size = 1, shuffle = False)定義模型
給定一組MRI腦掃描I = {I1,…In}及其對應的分割S = {S1,…Sn},我們想要學習一個函數fseg: I -> S。我們將這個函數表示為F-CNN模型,稱為QuickNAT:
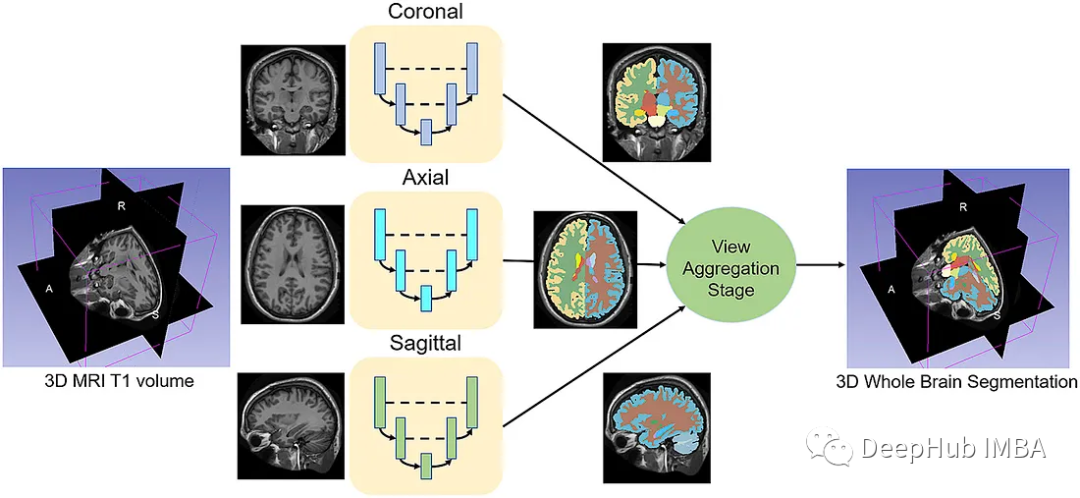
QuickNAT由三個二維f - cnn組成,分別在coronal, axial, sagittal視圖上操作,然后通過聚合步驟推斷最終的分割結果,該分割結果由三個網絡的概率圖組合而成。每個F-CNN都有一個編碼器/解碼器架構,其中有4個編碼器和4個解碼器,并由瓶頸層分隔。最后一層是帶有softmax的分類器塊。該架構還包括每個編碼器/解碼器塊內的殘差鏈接。
class QuickNat(nn.Module):
"""
A PyTorch implementation of QuickNAT
"""
def __init__(self, params):
"""
:param params: {'num_channels':1,
'num_filters':64,
'kernel_h':5,
'kernel_w':5,
'stride_conv':1,
'pool':2,
'stride_pool':2,
'num_classes':28
'se_block': False,
'drop_out':0.2}
"""
super(QuickNat, self).__init__()
# from monai.networks.blocks import squeeze_and_excitation as se
# self.cSE = ChannelSELayer(num_channels, reduction_ratio)
# self.encode1 = sm.EncoderBlock(params, se_block_type=se.SELayer.CSSE)
# params["num_channels"] = params["num_filters"]
# self.encode2 = sm.EncoderBlock(params, se_block_type=se.SELayer.CSSE)
# self.encode3 = sm.EncoderBlock(params, se_block_type=se.SELayer.CSSE)
# self.encode4 = sm.EncoderBlock(params, se_block_type=se.SELayer.CSSE)
# self.bottleneck = sm.DenseBlock(params, se_block_type=se.SELayer.CSSE)
# params["num_channels"] = params["num_filters"] * 2
# self.decode1 = sm.DecoderBlock(params, se_block_type=se.SELayer.CSSE)
# self.decode2 = sm.DecoderBlock(params, se_block_type=se.SELayer.CSSE)
# self.decode3 = sm.DecoderBlock(params, se_block_type=se.SELayer.CSSE)
# self.decode4 = sm.DecoderBlock(params, se_block_type=se.SELayer.CSSE)
# self.encode1 = EncoderBlock(params, se_block_type=se.ChannelSELayer)
self.encode1 = EncoderBlock(params, se_block_type=se.SELayer.CSSE)
params["num_channels"] = params["num_filters"]
self.encode2 = EncoderBlock(params, se_block_type=se.SELayer.CSSE)
self.encode3 = EncoderBlock(params, se_block_type=se.SELayer.CSSE)
self.encode4 = EncoderBlock(params, se_block_type=se.SELayer.CSSE)
self.bottleneck = DenseBlock(params, se_block_type=se.SELayer.CSSE)
params["num_channels"] = params["num_filters"] * 2
self.decode1 = DecoderBlock(params, se_block_type=se.SELayer.CSSE)
self.decode2 = DecoderBlock(params, se_block_type=se.SELayer.CSSE)
self.decode3 = DecoderBlock(params, se_block_type=se.SELayer.CSSE)
self.decode4 = DecoderBlock(params, se_block_type=se.SELayer.CSSE)
params["num_channels"] = params["num_filters"]
self.classifier = ClassifierBlock(params)
def forward(self, input):
"""
:param input: X
:return: probabiliy map
"""
e1, out1, ind1 = self.encode1.forward(input)
e2, out2, ind2 = self.encode2.forward(e1)
e3, out3, ind3 = self.encode3.forward(e2)
e4, out4, ind4 = self.encode4.forward(e3)
bn = self.bottleneck.forward(e4)
d4 = self.decode4.forward(bn, out4, ind4)
d3 = self.decode1.forward(d4, out3, ind3)
d2 = self.decode2.forward(d3, out2, ind2)
d1 = self.decode3.forward(d2, out1, ind1)
prob = self.classifier.forward(d1)
return prob
def enable_test_dropout(self):
"""
Enables test time drop out for uncertainity
:return:
"""
attr_dict = self.__dict__["_modules"]
for i in range(1, 5):
encode_block, decode_block = (
attr_dict["encode" + str(i)],
attr_dict["decode" + str(i)],
)
encode_block.drop_out = encode_block.drop_out.apply(nn.Module.train)
decode_block.drop_out = decode_block.drop_out.apply(nn.Module.train)
@property
def is_cuda(self):
"""
Check if model parameters are allocated on the GPU.
"""
return next(self.parameters()).is_cuda
def save(self, path):
"""
Save model with its parameters to the given path. Conventionally the
path should end with '*.model'.
Inputs:
- path: path string
"""
print("Saving model... %s" % path)
torch.save(self.state_dict(), path)
def predict(self, X, device=0, enable_dropout=False):
"""
Predicts the output after the model is trained.
Inputs:
- X: Volume to be predicted
"""
self.eval()
print("tensor size before transformation", X.shape)
if type(X) is np.ndarray:
# X = torch.tensor(X, requires_grad=False).type(torch.FloatTensor)
X = (
torch.tensor(X, requires_grad=False)
.type(torch.FloatTensor)
.cuda(device, non_blocking=True)
)
elif type(X) is torch.Tensor and not X.is_cuda:
X = X.type(torch.FloatTensor).cuda(device, non_blocking=True)
print("tensor size ", X.shape)
if enable_dropout:
self.enable_test_dropout()
with torch.no_grad():
out = self.forward(X)
max_val, idx = torch.max(out, 1)
idx = idx.data.cpu().numpy()
prediction = np.squeeze(idx)
print("prediction shape", prediction.shape)
del X, out, idx, max_val
return prediction損失函數
神經網絡的訓練需要一個損失函數來計算模型誤差。訓練的目標是最小化預測輸出和目標輸出之間的損失。我們的模型使用Dice Loss 和Weighted Logistic Loss的聯合損失函數進行優化,其中權重補償數據中的高類不平衡,并鼓勵正確分割解剖邊界。
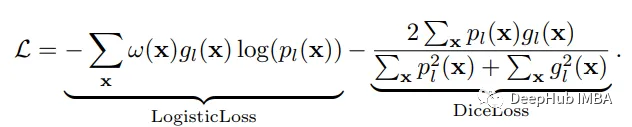
優化器
優化算法允許我們繼續更新模型的參數并最小化損失函數的值,我們設置了以下的超參數:
學習率:初始設置為0.1,10次后降低1階。這可以通過學習率調度器來實現。
權重衰減:0.0001。
批量大小:1。
動量:設置為0.95的高值,以補償由于小批量大小而產生的噪聲梯度。
訓練網絡
現在可以訓練模型了。對于QuickNAT需要在3個(coronal, axial, sagittal)2d切片上訓練3個模型。然后再聚合步驟中組合三個模型的概率生成最終結果,但是本文中只演示在coronal視圖的2D切片上訓練一個F-CNN模型,因為其他兩個與之類似。
num_epochs = 20
start_epoch = 1
val_interval = 1
train_loss_epoch_values = []
val_loss_epoch_values = []
best_ds_mean = -1
best_ds_mean_epoch = -1
ds_mean_train_values = []
ds_mean_val_values = []
# ds_LCE_values = []
# ds_LWM_values = []
# ds_LCC_values = []
print("START TRAINING. : model name = ", "quicknat")
for epoch in range(start_epoch, num_epochs):
print("==== Epoch ["+ str(epoch) + " / "+ str(num_epochs)+ "] DONE ====")
checkpoint_name = CHECKPOINT_DIR + "/checkpoint_epoch_" + str(epoch) + "." + CHECKPOINT_EXTENSION
print(checkpoint_name)
state = {
"epoch": epoch,
"arch": "quicknat",
"state_dict": model_coronal.state_dict(),
"optimizer": optimizer.state_dict(),
"scheduler": scheduler.state_dict(),
}
save_checkpoint(state = state, filename = checkpoint_name)
print("\n==== Epoch [ %d / %d ] START ====" % (epoch, num_epochs))
steps_per_epoch = len(train_dataset_coronal_subset) / train_loader_coronal_subset.batch_size
model_coronal.train()
train_loss_epoch = 0
val_loss_epoch = 0
step = 0
predictions_train = []
labels_train = []
predictions_val = []
labels_val = []
for i_batch, sample_batched in enumerate(train_loader_coronal_subset):
inputs = sample_batched['image'].type(torch.FloatTensor)
labels = sample_batched['label'].type(torch.LongTensor)
# print(f"Train Input Shape: {inputs.shape}")
labels = labels.squeeze(1)
_img_channels, _img_height, _img_width = labels.shape
encoded_label= np.zeros((_img_height, _img_width, 1)).astype(int)
for j, cls in enumerate(MaskColorMap):
encoded_label[np.all(labels == cls.value, axis = 0)] = j
labels = encoded_label
labels = torch.from_numpy(labels)
labels = torch.permute(labels, (2, 1, 0))
# print(f"Train Label Shape: {labels.shape}")
# plt.title("Train Label")
# plt.imshow(labels[0, :, :])
# plt.show()
optimizer.zero_grad()
outputs = model_coronal(inputs)
loss = loss_function(outputs, labels)
loss.backward()
optimizer.step()
scheduler.step()
with torch.no_grad():
_, batch_output = torch.max(outputs, dim = 1)
# print(f"Train Prediction Shape: {batch_output.shape}")
# plt.title("Train Prediction")
# plt.imshow(batch_output[0, :, :])
# plt.show()
predictions_train.append(batch_output.cpu())
labels_train.append(labels.cpu())
train_loss_epoch += loss.item()
print(f"{step}/{len(train_dataset_coronal_subset) // train_loader_coronal_subset.batch_size}, Training_loss: {loss.item():.4f}")
step += 1
predictions_train_arr, labels_train_arr = torch.cat(predictions_train), torch.cat(labels_train)
# print(predictions_train_arr.shape)
dice_metric(predictions_train_arr, labels_train_arr)
ds_mean_train = dice_metric.aggregate().item()
ds_mean_train_values.append(ds_mean_train)
dice_metric.reset()
train_loss_epoch /= step
train_loss_epoch_values.append(train_loss_epoch)
print(f"Epoch {epoch + 1} Train Average Loss: {train_loss_epoch:.4f}")
if (epoch + 1) % val_interval == 0:
model_coronal.eval()
step = 0
with torch.no_grad():
for i_batch, sample_batched in enumerate(val_loader_coronal_subset):
inputs = sample_batched['image'].type(torch.FloatTensor)
labels = sample_batched['label'].type(torch.LongTensor)
# print(f"Val Input Shape: {inputs.shape}")
labels = labels.squeeze(1)
integer_encoded_labels = []
_img_channels, _img_height, _img_width = labels.shape
encoded_label= np.zeros((_img_height, _img_width, 1)).astype(int)
for j, cls in enumerate(MaskColorMap):
encoded_label[np.all(labels == cls.value, axis = 0)] = j
labels = encoded_label
labels = torch.from_numpy(labels)
labels = torch.permute(labels, (2, 1, 0))
# print(f"Val Label Shape: {labels.shape}")
# plt.title("Val Label")
# plt.imshow(labels[0, :, :])
# plt.show()
val_outputs = model_coronal(inputs)
val_loss = loss_function(val_outputs, labels)
predicted = torch.argmax(val_outputs, dim = 1)
# print(f"Val Prediction Shape: {predicted.shape}")
# plt.title("Val Prediction")
# plt.imshow(predicted[0, :, :])
# plt.show()
predictions_val.append(predicted)
labels_val.append(labels)
val_loss_epoch += val_loss.item()
print(f"{step}/{len(val_dataset_coronal_subset) // val_loader_coronal_subset.batch_size}, Validation_loss: {val_loss.item():.4f}")
step += 1
predictions_val_arr, labels_val_arr = torch.cat(predictions_val), torch.cat(labels_val)
dice_metric(predictions_val_arr, labels_val_arr)
# dice_metric_batch(predictions_val_arr, labels_val_arr)
ds_mean_val = dice_metric.aggregate().item()
ds_mean_val_values.append(ds_mean_val)
# ds_mean_val_batch = dice_metric_batch.aggregate()
# ds_LCE = ds_mean_val_batch[0].item()
# ds_LCE_values.append(ds_LCE)
# ds_LWM = ds_mean_val_batch[1].item()
# ds_LWM_values.append(ds_LWM)
# ds_LCC = ds_mean_val_batch[2].item()
# ds_LCC_values.append(ds_LCC)
dice_metric.reset()
# dice_metric_batch.reset()
if ds_mean_val > best_ds_mean:
best_ds_mean = ds_mean_val
best_ds_mean_epoch = epoch + 1
torch.save(model_coronal.state_dict(), os.path.join(BESTMODEL_DIR, "best_metric_model_coronal.pth"))
print("Saved new best metric model coronal")
print(
f"Current Epoch: {epoch + 1} Current Mean Dice score is: {ds_mean_val:.4f}"
f"\nBest Mean Dice score: {best_ds_mean:.4f} "
# f"\nMean Dice score Left Cerebral Exterior: {ds_LCE:.4f} Mean Dice score Left White Matter: {ds_LWM:.4f} Mean Dice score Left Cerebral Cortex: {ds_LCC:.4f} "
f"at Epoch: {best_ds_mean_epoch}"
)
val_loss_epoch /= step
val_loss_epoch_values.append(val_loss_epoch)
print(f"Epoch {epoch + 1} Average Validation Loss: {val_loss_epoch:.4f}")
print("FINISH.")代碼也是傳統的Pytorch的訓練步驟,就不詳細解釋了
繪制損失和精度曲線
訓練曲線表示模型的學習情況,驗證曲線表示模型泛化到未見實例的情況。我們使用matplotlib來繪制圖形。還可以使用TensorBoard,它使理解和調試深度學習程序變得更容易,并且是實時的。
epoch = range(1, num_epochs + 1)
# Plot Loss Curves
plt.figure(figsize=(18, 6))
plt.subplot(1, 3, 1)
plt.plot(epoch, train_loss_epoch_values, label='Training Loss')
plt.plot(epoch, val_loss_epoch_values, label='Validation Loss')
plt.title('Training and Validation Loss')
plt.xlabel('Epoch')
plt.legend()
plt.figure()
plt.show()
# Plot Train Dice Coefficient Curve
plt.figure(figsize=(18, 6))
plt.subplot(1, 3, 2)
x = [(i + 1) for i in range(len(ds_mean_train_values))]
plt.plot(x, ds_mean_train_values, 'blue', label = 'Train Mean Dice Score')
plt.title("Training Mean Dice Coefficient")
plt.xlabel('Epoch')
plt.ylabel('Mean Dice Score')
plt.show()
# Plot Validation Dice Coefficient Curve
plt.figure(figsize=(18, 6))
plt.subplot(1, 3, 3)
x = [(i + 1) for i in range(len(ds_mean_val_values))]
plt.plot(x, ds_mean_val_values, 'orange', label = 'Validation Mean Dice Score')
plt.title("Validation Mean Dice Coefficient")
plt.xlabel('Epoch')
plt.ylabel('Mean Dice Score')
plt.show()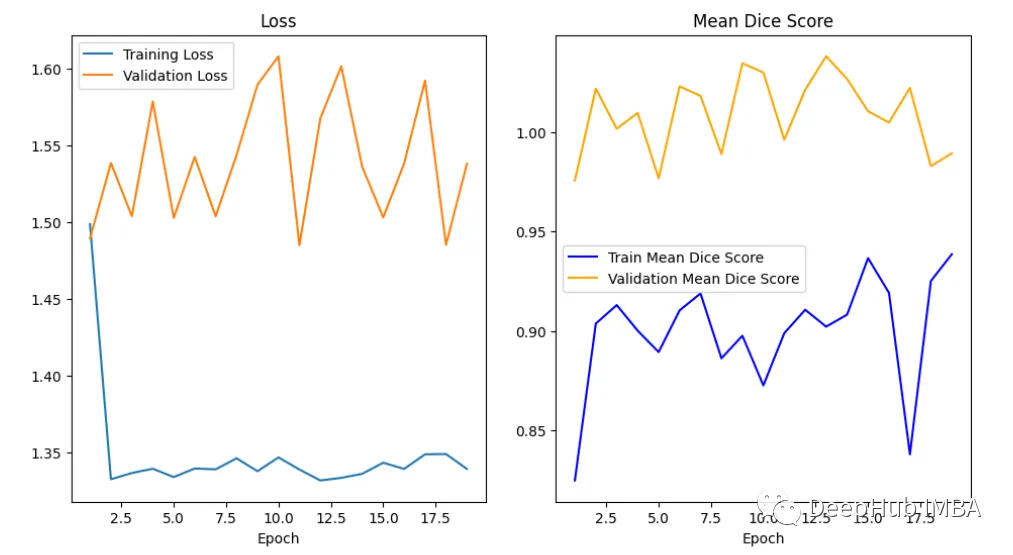
在曲線中,我們可以看到模型是過擬合的,因為驗證損失上升而訓練損失下降。這是深度學習算法中一個常見的陷阱,其中模型最終會記住訓練數據,而無法對未見過的數據進行泛化。
避免過度擬合的技巧:
- 用更多的數據進行訓練:更大的數據集可以減少過擬合。
- 數據增強:如果我們不能收集更多的數據,我們可以應用數據增強來人為地增加數據集的大小。
- 添加正則化:正則化是一種限制我們的網絡學習過于復雜的模型的技術,因此可能會過度擬合。
評估網絡
我們如何度量模型的性能?一個成功的預測是一個最大限度地擴大預測和真實之間的重疊。
這一目標的兩個相關但不同的指標是Dice和Intersection / Union (IoU)系數,后者也被稱為Jaccard系數。兩個指標都在0(無重疊)和1(完全重疊)之間。
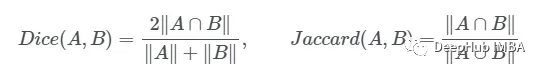
這兩種指標都可以用于類似的情況,但是區別在于Dice Score傾向于平均表現,而IoU則幫助你理解最壞情況下的表現。
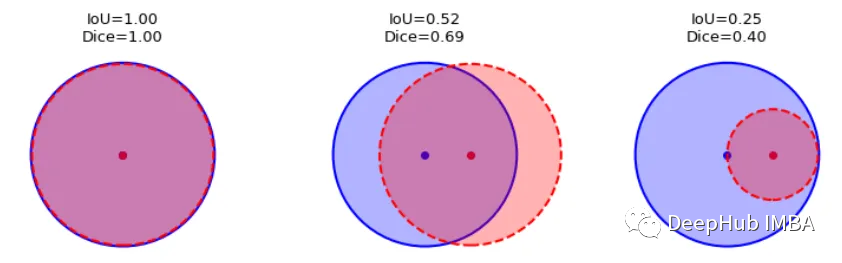
我們可以逐個類地檢查度量標準,或者取所有類的平均值。這里將使用monai.metrics.DiceMetric來計算分數。一個更通用的方法是使用torchmetrics,但是因為這里使用了monai框架,所以就直接使用它內置的函數了。
我們可以看到Dice得分曲線的行為相當不尋常。主要是因為驗證平均Dice得分高于1,這是不可能的,因為這個度量是在0和1之間。我們無法確定這種行為的主要原因,但我們建議在多類問題中為每個類單獨提供度量計算,并始終提供可視化示例以進行可視化評估。
結果分析
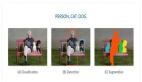
最后我們要看看模型是如何推廣到未知數據的這個模型預測的幾乎所有東西都是左腦白質,一些像素是左腦皮層。盡管它的預測似乎是正確的,但仍有很大的改進空間,因為我們的模型太小了,可以選擇更深的模型獲得更好的效果。
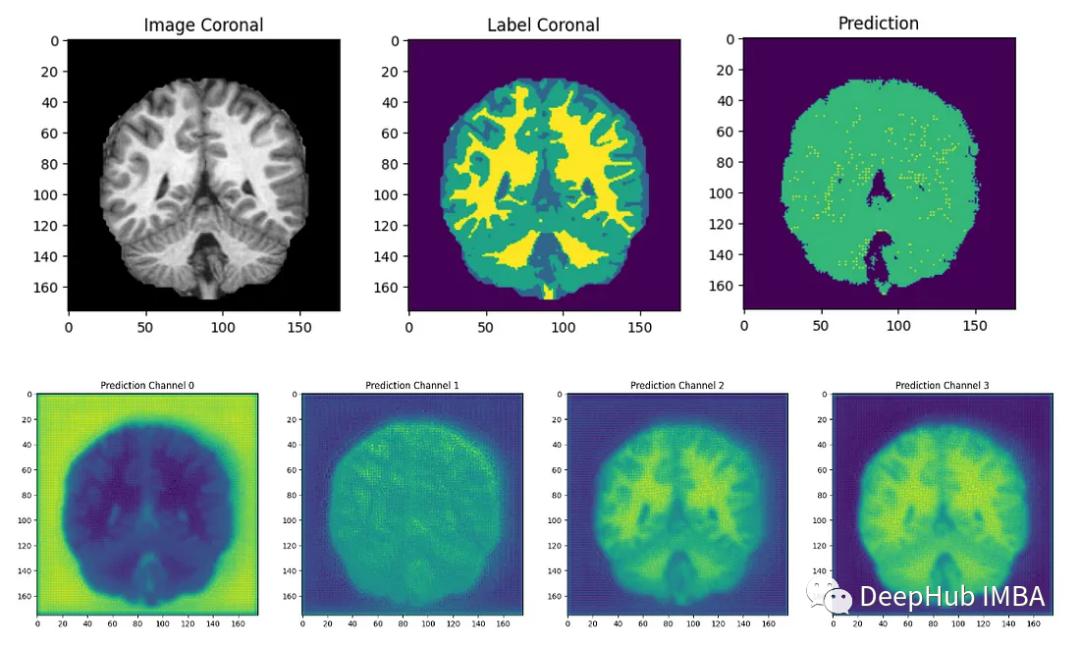
總結
在本文中,我們介紹了如何訓練QuickNAT來完成具有挑戰性的大腦分割任務。我們盡可能遵循作者在他們的研究論文中解釋的學習策略,這是本教程為了方便演示只在最簡單的步驟上進行了演示,文本的完整代碼:
https://github.com/inesdv26/Brain-Segmentation