昇思MindSpore再突破:蛋白質結構預測訓練推理全流程開源,助力生物醫藥發展
近日,昇思MindSpore與昌平實驗室、北京大學生物醫學前沿創新中心(BIOPIC)和化學與分子工程學院、深圳灣實驗室高毅勤教授課題組及鵬城實驗室陳杰團隊基于全場景AI框架昇思MindSpore實現AlphaFold2蛋白質結構訓練。繼2021年11月發布推理工具后,本次訓練意味著國產AI框架具備了強大的AI for Science底層軟件能力,同時也為相關科研工作者提供新的選擇。該聯合工作依托鵬城云腦II 昇騰AI 集群進行,單步迭代性能提升超過60%,TM-score達85分(國際權威評測數據集CASP14)。相關訓練代碼已在昇思MindSpore社區開源,后續也會在Openl啟智社區進行開源并定期擴展與維護。
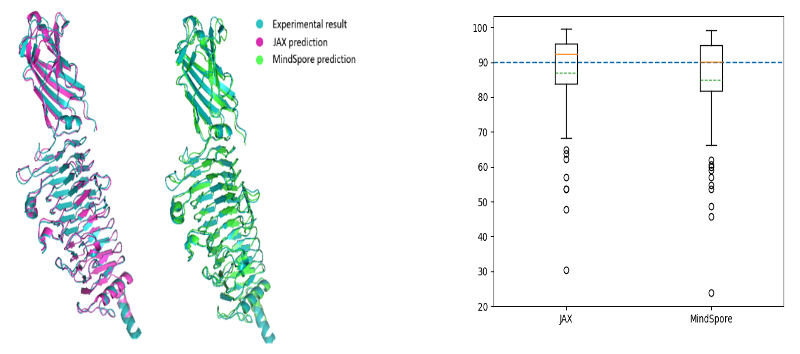
T1052-D1 預測結構圖(左)CASP14 87 targets TM-score 對比(右)
蛋白質結構預測是獲得蛋白質功能結構和構象的過程,近半個世紀以來,這一問題一直被譽為“21世紀的生物物理學”最重要的課題之一。在過去,因蛋白質構象數量巨大,計算過程復雜,通過AI來對蛋白質結構進行預測一直未能取得實質性突破,獲取蛋白質空間結構的方法仍然以冷凍電鏡、X-ray等實驗技術為主,單個蛋白質的觀測成本高達數月及數百萬人民幣。直至AlphaFold2的出現,使得這一問題迎來新的曙光。AlphaFold2憑借其接近實驗精度的成績取得CASP14蛋白質空間結構預測比賽的榜首,這一成就也被Nature譽為“前所未有的進步”。
2021年7月DeepMind宣布對AlphaFold2的推理代碼進行開源,昇思與高毅勤課題組第一時間對其進行了復現及優化,并于同年11月開源了基于昇思MindSpore的推理工具,效率同比提升2-3倍。由于開源范圍僅限推理,相關從業者無法基于此進行優化,因此許多團隊積極地投入訓練過程的復現。AlphaFold2模型本身存在內存需求大,數據處理繁瑣,控制編譯復雜等特點,對基礎AI框架存在著巨大挑戰。
近期,昇思MindSpore聯合高毅勤課題組、鵬城實驗室陳杰團隊全面打通AlphaFold2的訓練。采用昇騰基礎軟硬件平臺后,在混合精度下,單步迭代時間由20秒縮短到12秒,性能提升超過60%。依托昇思MindSpore內存復用能力, 訓練序列長度由384提升至512。
為了盡可能客觀地評估訓練結果,昇思MindSpore選取了AlphaFold2論文附錄中提到的87條驗證集進行驗證,平均TM-score達到85分,基本持平AlphaFold2。
昇思MindSpore對蛋白質結構預測訓練推理的支持填補了國產AI軟硬件的空白。在訓練精度接近AlphaFold2的基礎上,昇思MindSpore將在算法、規模和軟硬件支持等方向上持續改進,并計劃開放共享訓練數據集供同仁使用。昇思MindSpore也期望與更多學術界和工業界伙伴合作,進一步提升模型精度、擴展應用場景。
代碼開源路徑:
https://gitee.com/mindspore/mindscience/tree/dev/MindSPONGE/mindsponge/fold
昇思MindSpore:
Gitee:https://gitee.com/mindspore/mindspore
Github:https://github.com/mindspore-ai/mindspore