首個大眾可用PyTorch版AlphaFold2復現(xiàn),哥大開源,star量破千
剛剛,哥倫比亞大學系統(tǒng)生物學助理教授 Mohammed AlQuraishi 在推特上宣布,他們從頭訓練了一個名為 OpenFold 的模型,該模型是 AlphaFold2 的可訓練 PyTorch 復現(xiàn)版本。Mohammed AlQuraishi 還表示,這是第一個大眾可用的 AlphaFold2 復現(xiàn)。

AlphaFold2 可以周期性地以原子精度預測蛋白質結構,在技術上利用多序列對齊和深度學習算法設計,并結合關于蛋白質結構的物理和生物學知識提升了預測效果。它實現(xiàn)了 2/3 蛋白質結構預測的卓越成績并在去年登上了《自然》雜志。更令人驚喜的是,DeepMind 團隊不僅開源了模型,還將 AlphaFold2 預測數(shù)據(jù)做成了免費開放的數(shù)據(jù)集。
然而,開源并不意味著能用、好用。其實,AlphaFold2 軟件系統(tǒng)的部署難度極大,并且對硬件的要求高、數(shù)據(jù)集下載周期長、占用空間大,每一條都讓普通開發(fā)者望而卻步。因此,開源社區(qū)一直在努力實現(xiàn) AlphaFold2 的可用版本。
這次哥倫比亞大學 Mohammed AlQuraishi 教授等人實現(xiàn)的 OpenFold 總訓練時間大約為 100000 A100 小時,但在大約 3000 小時內就達到了 90% 的準確率。

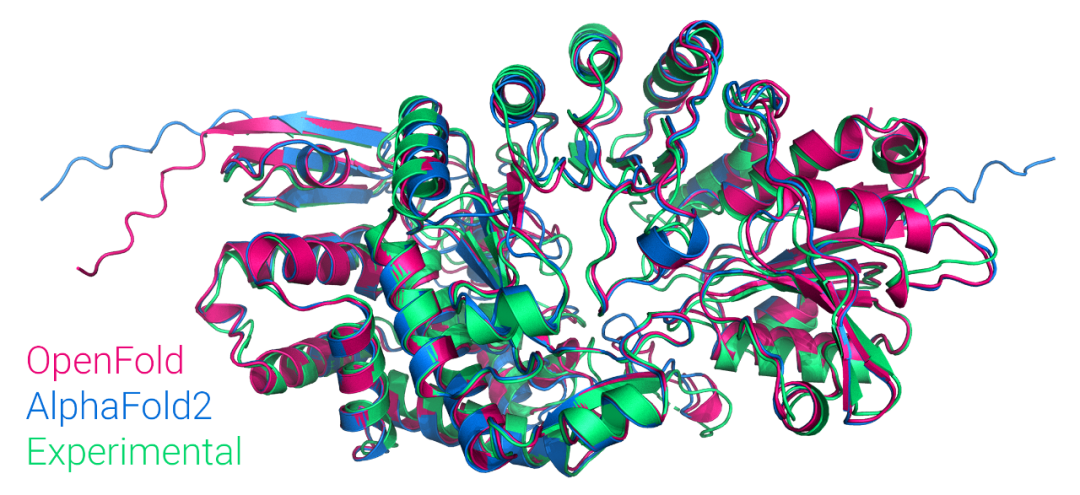
OpenFold 與原版 AlphaFold2 的準確率相當,甚至略勝一籌,可能因為 OpenFold 的訓練集更大一點:
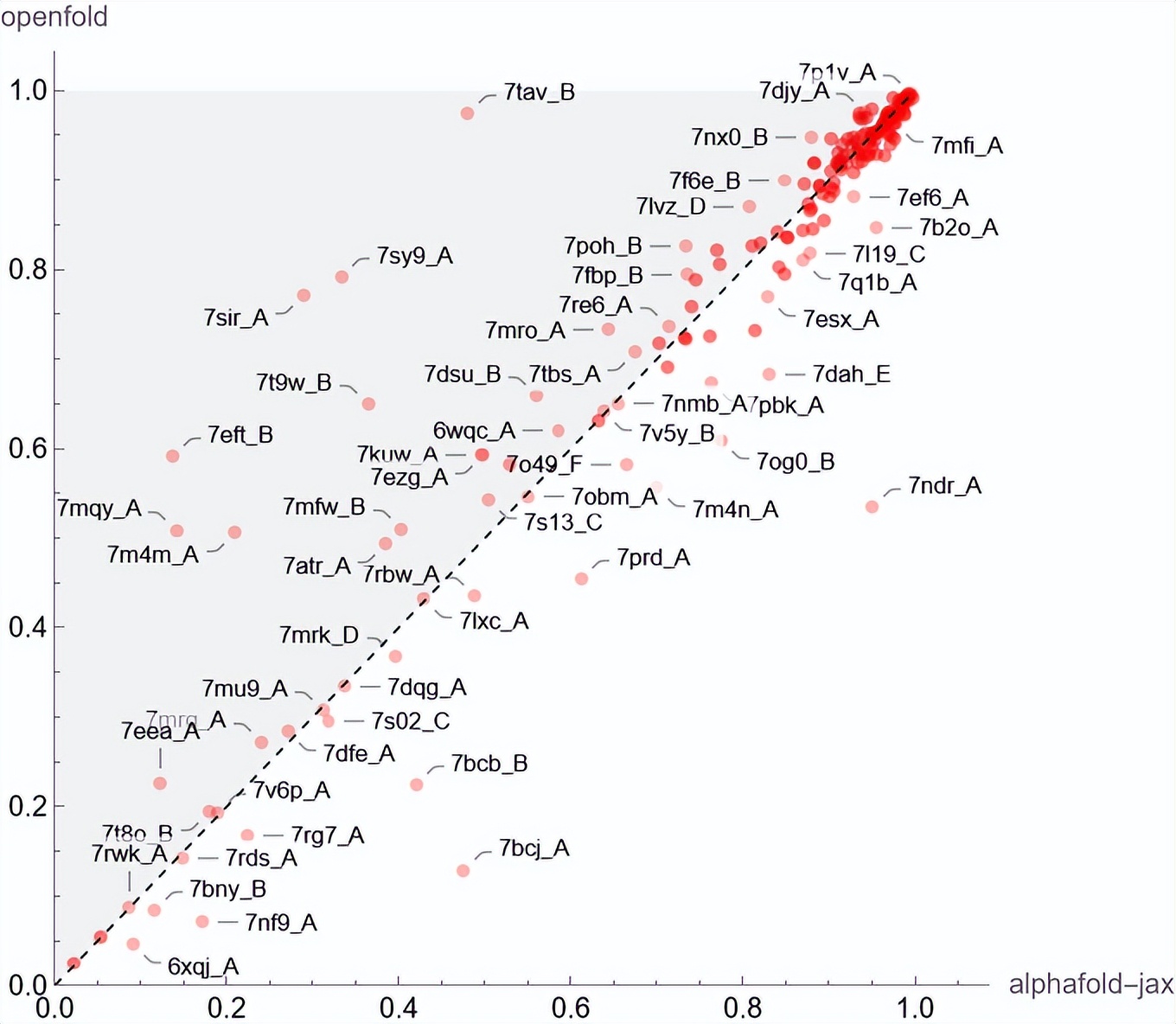
OpenFold 的主要優(yōu)勢是推理速度顯著提升,對于較短的蛋白質序列,OpenFold 的推理速度可以達到 AlphaFold2 的兩倍。另外,由于使用自定義的 CUDA 內核,OpenFold 使用更少的內存就能推理更長的蛋白質序列。
OpenFold 介紹
OpenFold 幾乎再現(xiàn)了原始開源推理代碼 (v2.0.1) 的所有功能,除了已趨于被淘汰的「模型集成」功能,該功能在 DeepMind 自己的消融測試中就表現(xiàn)不佳。
無論是否有 DeepSpeed,OpenFold 都能以全精度或 bfloat16 進行訓練。為了實現(xiàn) AlphaFold2 的原始性能,該團隊從頭開始訓練 OpenFold,現(xiàn)已公開發(fā)布了模型權重和訓練數(shù)據(jù)。其中,訓練數(shù)據(jù)包含大約 400000 份 MSA 和 PDB70 模板文件。OpenFold 還支持使用 AlphaFold 的官方參數(shù)進行蛋白質推理。
與其他實現(xiàn)相比,OpenFold 具有以下優(yōu)點:
- 短序列推理:加快了在 GPU 上推理少于 1500 個氨基酸殘基的鏈的速度;
- 長序列推理:通過該研究實現(xiàn)的低記憶注意力(low-memory attention)對極長鏈進行推理,OpenFold 可以在單個 A100 上預測 超過 4000 個殘基的序列結構,借助 CPU offload 甚至可以預測更長的序列;
- 內存高效在訓練和推理期間,在 FastFold 內核基礎上修改的自定義 CUDA 注意力內核,使用的 GPU 內存分別比等效的 FastFold 和現(xiàn)有的 PyTorch 實現(xiàn)少 4 倍和 5 倍;
- 高效對齊腳本:該團隊使用原始 AlphaFold HHblits/JackHMMER pipeline 或帶有 MMseqs2 的 ColabFold,已經(jīng)生成了數(shù)百萬個對齊。
Linux 系統(tǒng)下的安裝與使用
開發(fā)團隊提供了一個在本地安裝 Miniconda、創(chuàng)建 conda 虛擬環(huán)境、安裝所有 Python 依賴項并下載有用資源的腳本,包括兩組模型參數(shù)。
運行以下命令:
scripts/install_third_party_dependencies.sh
使用如下命令激活環(huán)境:
source scripts/activate_conda_env.sh
停用命令:
source scripts/deactivate_conda_env.sh
在激活環(huán)境下,編譯 OpenFold 的 CUDA 內核
python3 setup.py install
在 / usr/bin 路徑下安裝 HH-suite:
# scripts/install_hh_suite.sh
使用如下命令可以下載用于訓練 OpenFold 和 AlphaFold 的數(shù)據(jù)庫:
bash scripts/download_data.sh data/
如果要使用一組 DeepMind 的預訓練參數(shù)對一個或多個序列進行推理,可以運行如下代碼:
python3 run_pretrained_openfold.py \
fasta_dir \
data/pdb_mmcif/mmcif_files/ \
--uniref90_database_path data/uniref90/uniref90.fasta \
--mgnify_database_path data/mgnify/mgy_clusters_2018_12.fa \
--pdb70_database_path data/pdb70/pdb70 \
--uniclust30_database_path data/uniclust30/uniclust30_2018_08/uniclust30_2018_08 \
--output_dir ./ \
--bfd_database_path data/bfd/bfd_metaclust_clu_complete_id30_c90_final_seq.sorted_opt \
--model_device "cuda:0" \
--jackhmmer_binary_path lib/conda/envs/openfold_venv/bin/jackhmmer \
--hhblits_binary_path lib/conda/envs/openfold_venv/bin/hhblits \
--hhsearch_binary_path lib/conda/envs/openfold_venv/bin/hhsearch \
--kalign_binary_path lib/conda/envs/openfold_venv/bin/kalign
--config_preset "model_1_ptm"
--openfold_checkpoint_path openfold/resources/openfold_params/finetuning_2_ptm.pt
更多細節(jié)請參見 GitHub:https://github.com/aqlaboratory/openfold
擴展閱讀:
- 高效預測幾乎所有人類蛋白質結構,AlphaFold 再登 Nature,數(shù)據(jù)庫全部免費開放
- 生物計算專家超細致解讀 AlphaFold2 論文:模型架構及應用
- DeepMind 開源的 AlphaFold 怎么用?打開 Colab 就能在線用