蛋白質側鏈預測新方法DiffPack:擴散模型也能精準預測側鏈構象!
蛋白質由氨基酸經脫水縮合反應形成,可折疊為復雜的 3D 空間結構,是生物體內執行各種功能(催化生物化學反應、傳遞信號)的主要分子,而其中蛋白質的側鏈結構對其功能至關重要。側鏈的化學性質和空間排列決定了蛋白質如何折疊,以及蛋白質如何與其他分子(包括其他蛋白質、DNA、RNA、小分子等)相互作用。這些相互作用可以是氫鍵、離子鍵、疏水相互作用和范德華力等。理解這些相互作用,可以幫助我們更好的尋找藥物與受體的結合位點,設計催化效果更強的酶。因此,準確預測蛋白質的側鏈構象對于理解蛋白質的功能和設計新的藥物至關重要。
傳統的蛋白質側鏈構象預測算法(如 RosettaPacker)通常通過在能量函數定義的概率分布中采樣得到蛋白質側鏈構象,此類算法通常依賴于人為設計的能量函數,受限于能量函數設計的精準度。另一方面,盡管以 AlphaFold2 為首的一系列深度學習模型在蛋白質結構預測方面取得了比較大的突破,但其對側鏈構象的預測通常依賴于端對端的學習,無法捕捉側鏈構象的整體分布,因此準確度上受到了一定的限制。
近日,來自 Mila 的唐建團隊提出了一種新的側鏈構象預測方法 DiffPack。該方法使用扭轉角空間中的自回歸擴散模型,與目前已知的最優方法相比,在 CASP13 和 CASP14 上取得了大幅提升實現了超過 10% 的提升,且僅需要 1/60 的參數量。此外,作者進一步驗證了 DiffPack 能夠給有效改進 AlphaFold2 的側鏈預測結果。
 圖片
圖片
- 論文鏈接:https://arxiv.org/abs/2306.01794
- 代碼鏈接:https://github.com/DeepGraphLearning/DiffPack
方法
過去的 AI 算法以端對端的方式從原子位置的均方根偏差 (RMSD)定義損失函數。盡管此類方法存在速度上的優勢,但他們通常被端對端的建模方式所限制,無法捕捉蛋白質側鏈構象的整體分布,傾向于生成側鏈的 「平均構象」。DiffPack 基于擴散模型提出了一種新穎的側鏈構象預測方法。通過對蛋白質側鏈構象加入噪聲并學習去噪過程,模型能夠更準確地學習到蛋白質側鏈構象的整體概率分布。
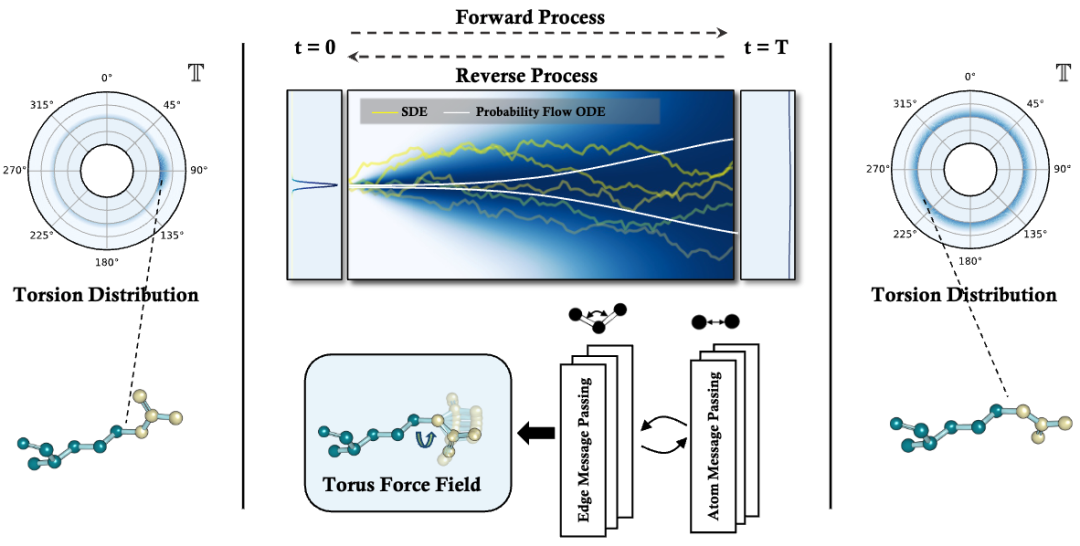
蛋白質側鏈的構象通常較為復雜,如果直接使用空間坐標的擴散過程建模會引入過多的自由度從而增加問題的復雜性。同時由于蛋白質的側鏈構象由至多四個扭轉角(χ1, χ2, χ3, χ4)構成,研究人員選擇將擴散過程定義在四個扭轉角構成的扭轉空間中,在前向擴散過程中引入非歐幾里得噪聲,同時通過針對蛋白質結構設計的圖神經網絡 GearNet 學習對四個扭轉角的逆向去噪過程。
 圖片
圖片
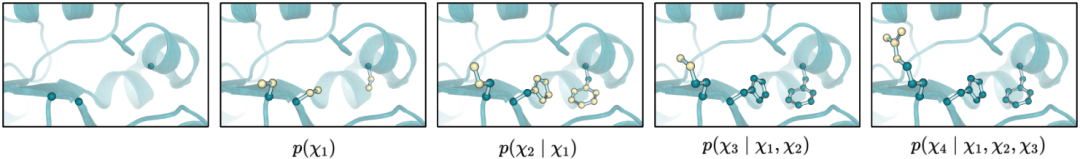
在實驗中,研究人員發現對四個扭轉角的聯合加噪過程會會產生偏差累積效應,從而降低預測的準確度。研究人員由此進一步提出了一種新穎的自回歸擴散模型,將四個扭轉角的聯合概率分布分解為一系列條件概率分布,在每一個概率分布中引入擴散模型。通過將擴散模型與自回歸過程結合,DiffPack 能夠以很小的模型規模實現高精度的側鏈構象預測。
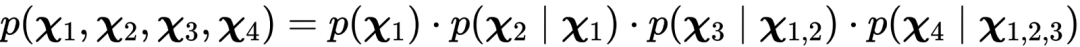
 圖片
圖片
實驗
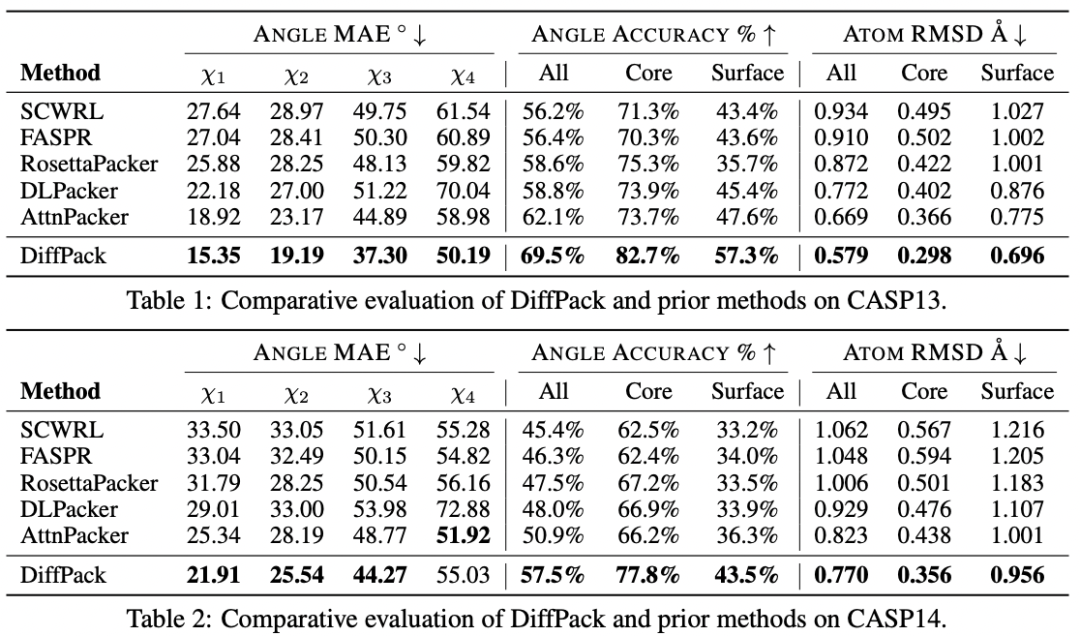
研究人員在包括天然主鏈結構和由 AlphaFold2 預測得到的非天然主鏈結構上進行了實驗。DiffPack 在預測蛋白質側鏈構象中超越了基于傳統能量函數的方法和其他端對端的深度學習模型。表 1 總結了在 CASP13 中的實驗結果,DiffPack 無論是在內部殘基(82.7%)和表面殘基(57.3%)上,都表現出了最高的角度精度。對于表面殘基,其精度比之前的最先進模型 AttnPacker 提高了 20.4%。
 圖片
圖片
同樣,DiffPack 在 CASP14 數據集的表現也優于其他方法(表 2),盡管沒有將損失函數直接定義在原子位置上,DiffPack 在內部殘基原子位置的均方根偏差精度上依然相比于之前的 SOTA 提高了 23%。
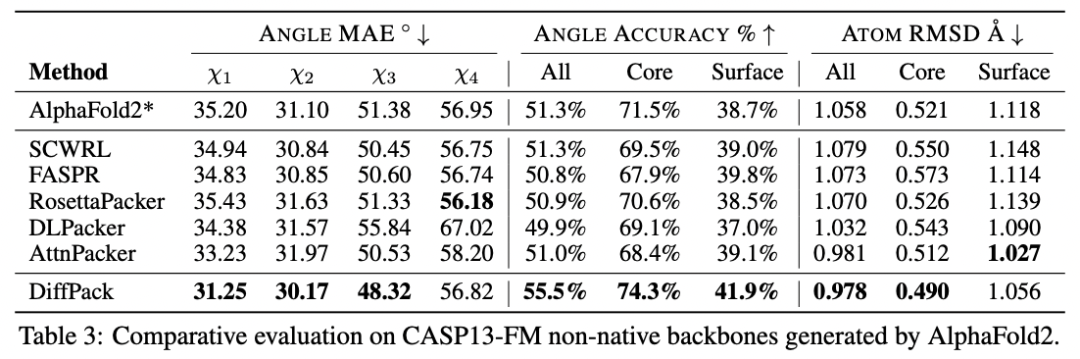
由于 AlphaFold2 等計算結構生物學技術的發展,當前人們的關注重心逐漸轉移到非天然主鏈結構的應用上。因此研究人員將 DiffPack 進一步應用到有 AlphaFold2 生成的非天然主鏈結構,表 3 給出了包括 AlphaFold2 自身的側鏈預測算法在內的不同算法在非天然主鏈結構測試集上的定量結果。DiffPack 在大多數指標上實現了最先進的水平,這說明了 DiffPack 可以進一步優化 AlphaFold2 預測的潛力。
 圖片
圖片
DiffPack 除了在對側鏈構象的準確度上優于其他方法,其模型的總參數量要顯著(3,043,363)少于此前的 SOTA 模型(208,098,163),這使得 DiffPack 成為預測側鏈構象的更具計算可行性和可擴展性的解決方案。
總結
- 研究人員基于擴散模型提出了一種新的針對蛋白質側鏈構象的預測算法 DiffPack,DiffPack 將扭轉空間中的擴散模型與自回歸過程結合,能夠更好地捕捉蛋白質側鏈構象分布。
- DiffPack 在天然主鏈結構與非天然主鏈結構的預測上都表現出了一定的優勢,同時其模型規模要顯著小于其他方法。






































