Meta AI開放6億+宏基因組蛋白質(zhì)結(jié)構(gòu)圖譜,150億語言模型用兩周完成
今年,DeepMind 公布了大約 2.2 億種蛋白質(zhì)的預(yù)測(cè)結(jié)構(gòu),它幾乎涵蓋了 DNA 數(shù)據(jù)庫中已知生物體的所有蛋白質(zhì)。現(xiàn)在,另一家科技巨頭 Meta 正在填補(bǔ)另一空白,微生物領(lǐng)域。
簡單來說,Meta 使用 AI 技術(shù)預(yù)測(cè)了約 6 億種蛋白質(zhì)結(jié)構(gòu),這些蛋白質(zhì)來自細(xì)菌和其他尚未被表征的微生物。團(tuán)隊(duì)負(fù)責(zé)人 Alexander Rives 表示:「這些蛋白質(zhì)是我們所知最少的結(jié)構(gòu),它們是非常神秘的蛋白質(zhì)。我認(rèn)為這些發(fā)現(xiàn)為深入了解生物學(xué)提供了潛力。」
通常,語言模型是在大量文本上進(jìn)行訓(xùn)練的。Meta 為了將語言模型應(yīng)用于蛋白質(zhì),Rives 及其同事將已知的蛋白質(zhì)序列作為輸入,這些蛋白質(zhì)由 20 種氨基酸組成,并用不同的字母表示。然后,該網(wǎng)絡(luò)在遮蔽一定比例氨基酸的情況下學(xué)會(huì)了自動(dòng)補(bǔ)全蛋白質(zhì)。
Meta 將這個(gè)網(wǎng)絡(luò)命名為 ESMFold。雖然 ESMFold 預(yù)測(cè)準(zhǔn)確性不如 AlphaFold,但在預(yù)測(cè)結(jié)構(gòu)方面,它比 AlphaFold 快約 60 倍。這一速度意味著可以將蛋白質(zhì)結(jié)構(gòu)預(yù)測(cè)擴(kuò)展到更大的數(shù)據(jù)庫。

- 論文地址:https://www.biorxiv.org/content/10.1101/2022.07.20.500902v2
- 項(xiàng)目地址:https://github.com/facebookresearch/esm
如今,作為測(cè)試,Meta 決定將他們的模型應(yīng)用于宏基因組 DNA 數(shù)據(jù)庫,這些 DNA 全部來自環(huán)境,包括土壤、海水、人類腸道、皮膚和其他微生物棲息地。Meta AI 宣布推出包含 6 億多個(gè)蛋白質(zhì)的 ESM 宏基因組圖譜(ESM Metagenomic Atlas),它是首個(gè)蛋白質(zhì)宇宙「暗物質(zhì)」的綜合視圖。這還是最大的高分辨率預(yù)測(cè)結(jié)構(gòu)數(shù)據(jù)庫,比任何現(xiàn)有的蛋白質(zhì)結(jié)構(gòu)數(shù)據(jù)庫都要大 3 倍,并且是第一個(gè)全面、大規(guī)模地涵蓋宏基因組蛋白質(zhì)的數(shù)據(jù)庫。
Meta 團(tuán)隊(duì)總共預(yù)測(cè)了超過 6.17 億個(gè)蛋白質(zhì)結(jié)構(gòu),只花了兩周的時(shí)間。Rives 說,預(yù)測(cè)是免費(fèi)的,任何人都可以使用,就像模型的底層代碼一樣。
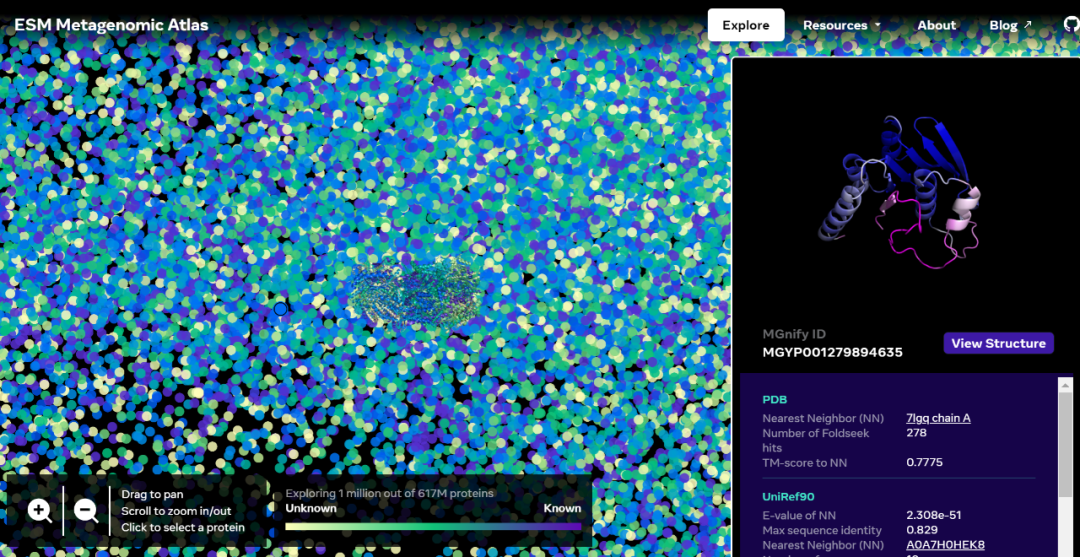
交互版本地址:https://esmatlas.com/explore?at=1%2C1%2C21.999999344348925
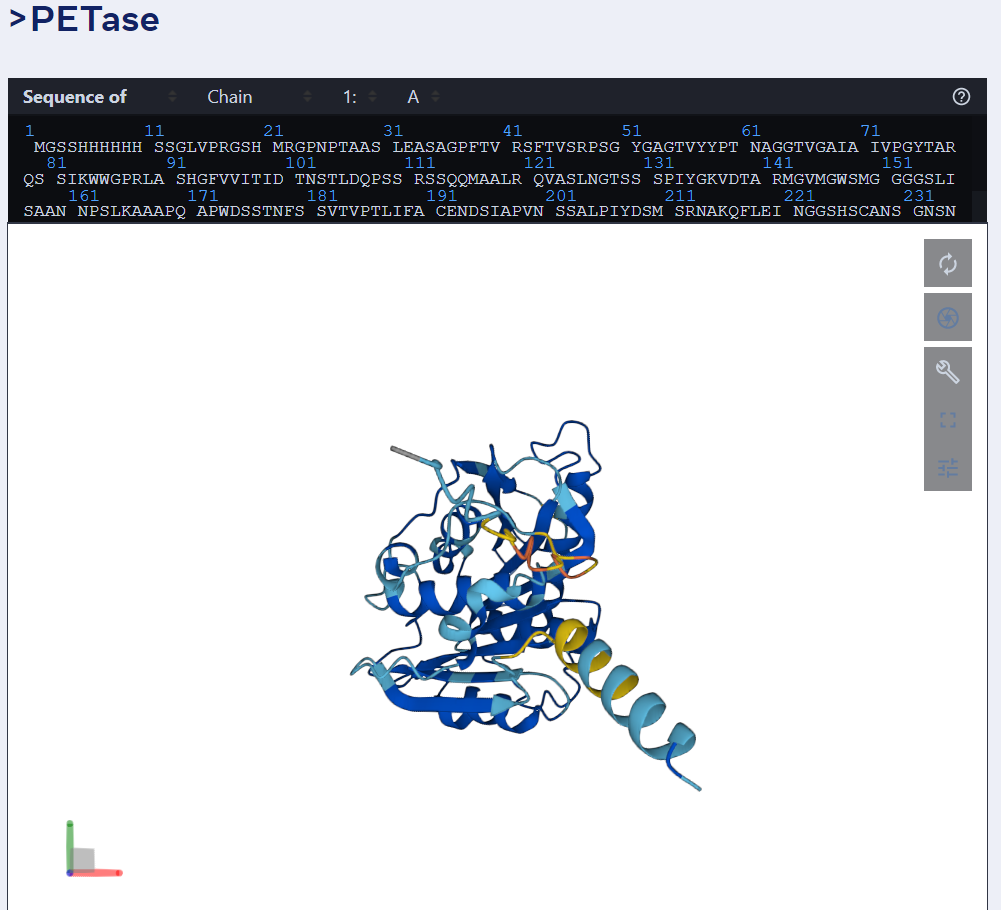
舉例而言,下圖為 ESMFold 對(duì) PET 酶的預(yù)測(cè)。
引言
眾所周知,蛋白質(zhì)作為復(fù)雜且動(dòng)態(tài)的分子,其由基因編碼,主要負(fù)責(zé)生命基本過程。蛋白質(zhì)在生物學(xué)中有著驚人作用。比如,人類眼睛中的視桿和視錐細(xì)胞可以感知光線,因而我們能看到外面的世界;構(gòu)成聽覺和觸覺基礎(chǔ)的分子傳感器;植物中把光能轉(zhuǎn)化為化學(xué)能的復(fù)雜分子;驅(qū)動(dòng)微生物和人類肌肉運(yùn)動(dòng)的「馬達(dá)」;分解塑料的酶;保護(hù)我們免受疾病的抗體,等等這些都是蛋白質(zhì)。
1998 年,來自威斯康辛大學(xué)植物病理學(xué)部門的 Jo Handelsman 首次提出宏基因組學(xué)(Metagenomics)這一概念,它是源于將來自環(huán)境中基因集可以在某種程度上當(dāng)成單個(gè)基因組研究分析的想法,而宏的英文正是 meta-,也翻譯為元。
宏基因組學(xué)揭示了數(shù)十億個(gè)對(duì)科學(xué)來說是新的蛋白質(zhì)序列,并首次編入由 NCBI、歐洲生物信息學(xué)研究所 (European Bioinformatics Institute) 和聯(lián)合基因組研究所 (Joint Genome Institute) 等公共項(xiàng)目編制的大型數(shù)據(jù)庫中。
Meta AI 開發(fā)的新的蛋白質(zhì)折疊方法,該方法利用大型語言模型,在宏基因組數(shù)據(jù)庫中(具有數(shù)億蛋白質(zhì))創(chuàng)建了首個(gè)全面的蛋白質(zhì)結(jié)構(gòu)視圖。Meta 發(fā)現(xiàn),相對(duì)于現(xiàn)有的 SOTA 蛋白質(zhì)結(jié)構(gòu)預(yù)測(cè)方法,語言模型可以將預(yù)測(cè)蛋白質(zhì)原子級(jí)三維結(jié)構(gòu)的速度提高 60 倍。這一進(jìn)展將有助于加速蛋白質(zhì)結(jié)構(gòu)理解的新時(shí)代,這是首次人類有可能了解基因測(cè)序技術(shù)正在編目的數(shù)十億蛋白質(zhì)的結(jié)構(gòu)。
解鎖隱藏的自然世界:宏基因組結(jié)構(gòu)空間的首個(gè)綜合視圖
我們知道,基因測(cè)序的進(jìn)步使得對(duì)數(shù)十億個(gè)宏基因組蛋白序列進(jìn)行編目成為可能。但是,通過實(shí)驗(yàn)確定數(shù)以億計(jì)蛋白質(zhì)的 3D 結(jié)構(gòu)遠(yuǎn)遠(yuǎn)超出了時(shí)間密集型實(shí)驗(yàn)室技術(shù)的范圍,例如 X 射線晶體學(xué),它可能需要數(shù)周乃至數(shù)年的時(shí)間來檢測(cè)單個(gè)蛋白質(zhì)。計(jì)算方式可以讓我們深入了解使用實(shí)驗(yàn)技術(shù)無法實(shí)現(xiàn)的宏基因組學(xué)蛋白質(zhì)。
ESM 宏基因組圖譜將使科學(xué)家能夠在數(shù)億蛋白質(zhì)的尺度上搜索和分析宏基因組蛋白質(zhì)的結(jié)構(gòu)。這可以幫助識(shí)別以前未被表征的結(jié)構(gòu),尋找遙遠(yuǎn)的進(jìn)化關(guān)系,并發(fā)現(xiàn)可用于醫(yī)學(xué)和其他應(yīng)用的新蛋白質(zhì)。
如下為一張包含數(shù)以萬計(jì)高置信度預(yù)測(cè)的圖譜,展示了與目前已知結(jié)構(gòu)的蛋白質(zhì)的相似性。并且,該圖像首次顯示了完全未知的蛋白質(zhì)結(jié)構(gòu)空間的更大區(qū)域。
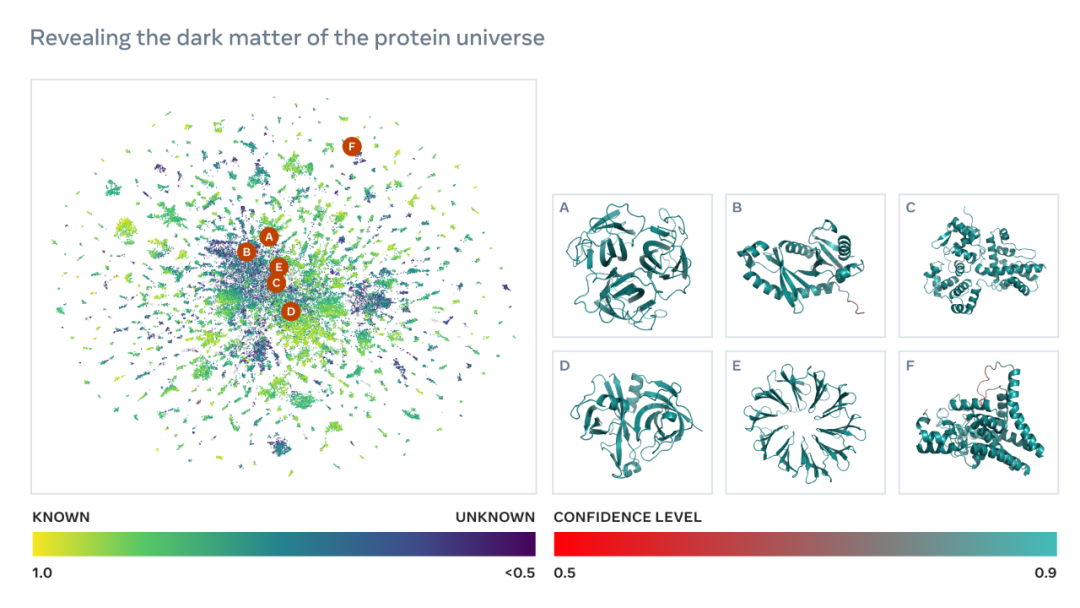
學(xué)習(xí)閱讀生物學(xué)語言
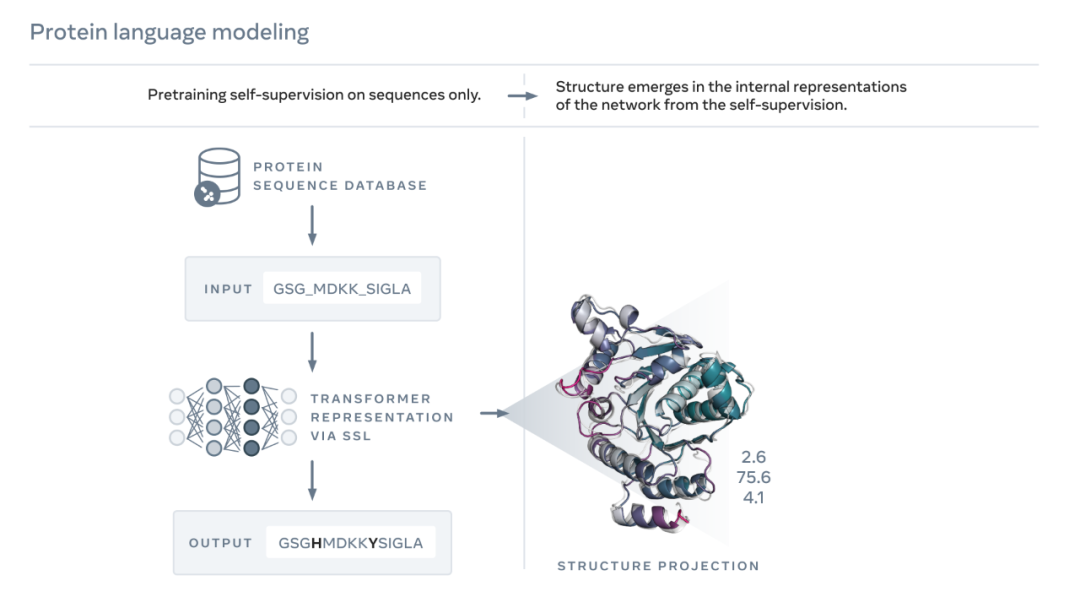
如下圖所示,ESM-2 語言模型經(jīng)過訓(xùn)練,可以預(yù)測(cè)進(jìn)化過程中被序列掩蓋的氨基酸。Meta AI 發(fā)現(xiàn),作為訓(xùn)練的結(jié)果,蛋白質(zhì)結(jié)構(gòu)的信息出現(xiàn)在該模型的內(nèi)部狀態(tài)中。這實(shí)在令人驚訝,因?yàn)樵撃P蛢H在序列上進(jìn)行了訓(xùn)練。
 ?
?
就像論文或信件的文本一樣,蛋白質(zhì)可以寫成字符序列。其中,每個(gè)字符對(duì)應(yīng) 20 種標(biāo)準(zhǔn)化學(xué)元素(氨基酸)中的一種,每種又具有不同的特性,它們是蛋白質(zhì)的構(gòu)建塊。這些構(gòu)建塊能夠以天文數(shù)字的不同方式組合在一起,例如對(duì)于由 200 個(gè)氨基酸組成的蛋白質(zhì),存在 20^200 個(gè)可能的序列,這要比可見宇宙中的原子數(shù)量還要多。每個(gè)序列都折疊成 3D 形狀(但并非所有序列都會(huì)折疊成連貫的結(jié)構(gòu),許多序列折疊成無序形式),正是這種形狀在很大程度上決定了蛋白質(zhì)的生物學(xué)功能。
學(xué)習(xí)閱讀這種生物學(xué)語言帶來了很大挑戰(zhàn)。雖然蛋白質(zhì)序列和文本段落都可以寫成字符,但它們之間存在著深刻而根本性的差異。蛋白質(zhì)序列描述了一個(gè)分子的化學(xué)結(jié)構(gòu),該分子根據(jù)物理定律折疊成復(fù)雜的 3D 形狀。
蛋白質(zhì)序列包含了傳遞蛋白質(zhì)折疊結(jié)構(gòu)信息的統(tǒng)計(jì)模式。舉例而言,如果一個(gè)蛋白質(zhì)中的兩個(gè)位置共同進(jìn)化,或者換言之,如果其中一個(gè)位置出現(xiàn)某種氨基酸,通常與另一個(gè)位置的某種氨基酸配對(duì),這可能意味著這兩個(gè)位置在折疊結(jié)構(gòu)中相互作用。這類似于拼圖游戲中的兩塊拼圖,進(jìn)化必須選擇在折疊結(jié)構(gòu)中拼合在一起的氨基酸。這又意味著我們通常可以通過觀察蛋白質(zhì)序列中的模式來推斷蛋白質(zhì)的結(jié)構(gòu)。
ESM 使用 AI 來學(xué)習(xí)閱讀這些模式。2019 年,Meta AI 提供證據(jù)證明語言模型學(xué)習(xí)了蛋白質(zhì)的特性,例如它們的結(jié)構(gòu)和功能。通過一種被稱為掩碼語言建模的自我監(jiān)督學(xué)習(xí)形式,Meta AI 在數(shù)百萬個(gè)天然蛋白質(zhì)的序列上訓(xùn)練了一個(gè)語言模型。使用這種方法,模型必須正確填寫文本段落中的空白,例如「To _ or not to , that is the _____」。
之后,Meta AI 訓(xùn)練了一個(gè)語言模型來填補(bǔ)蛋白質(zhì)序列中的空白。他們發(fā)現(xiàn),蛋白質(zhì)結(jié)構(gòu)和功能的信息在這一訓(xùn)練中浮現(xiàn)了出來。2020 年,Meta 發(fā)布了一個(gè) SOTA 蛋白質(zhì)語言模型 ESM1b,用于各種應(yīng)用,包括幫助科學(xué)家預(yù)測(cè) COVID-19 的演變以及發(fā)現(xiàn)疾病的遺傳原因。
現(xiàn)在,Meta AI 擴(kuò)展了這種方法,用來創(chuàng)建下一代蛋白質(zhì)語言模型 ESM-2,它的參數(shù)為 150 億,是迄今為止最大的蛋白質(zhì)語言模型。他們發(fā)現(xiàn),當(dāng)模型參數(shù)從 800 萬放大到 150 億時(shí),內(nèi)部表示中會(huì)出現(xiàn)信息,從而能夠以原子分辨率進(jìn)行 3D 結(jié)構(gòu)預(yù)測(cè)。
將蛋白質(zhì)折疊實(shí)現(xiàn)數(shù)量級(jí)加速
在下圖中,隨著模型的擴(kuò)大,高分辨率的蛋白質(zhì)結(jié)構(gòu)出現(xiàn)。同時(shí)隨著模型的縮放,蛋白質(zhì)結(jié)構(gòu)的原子分辨率圖像中會(huì)出現(xiàn)新的細(xì)節(jié)。
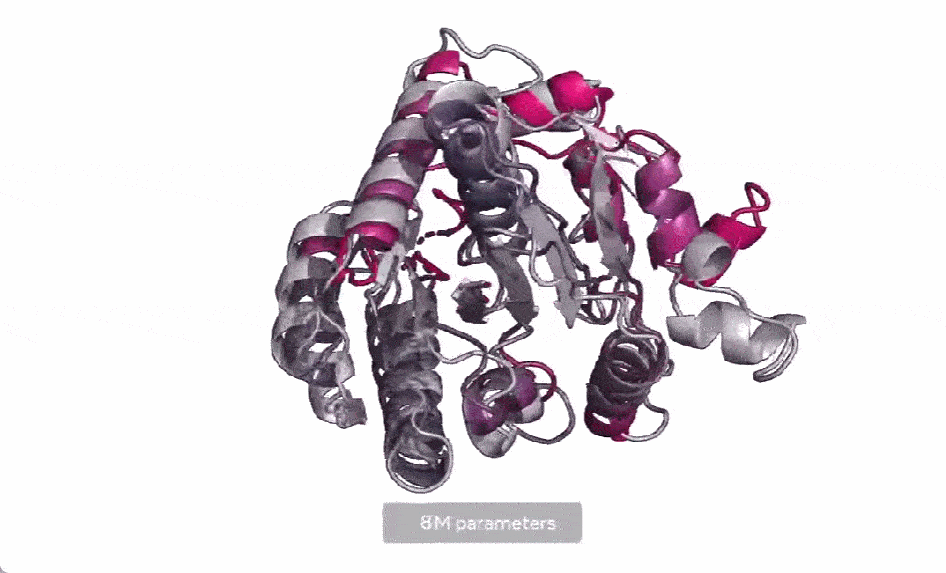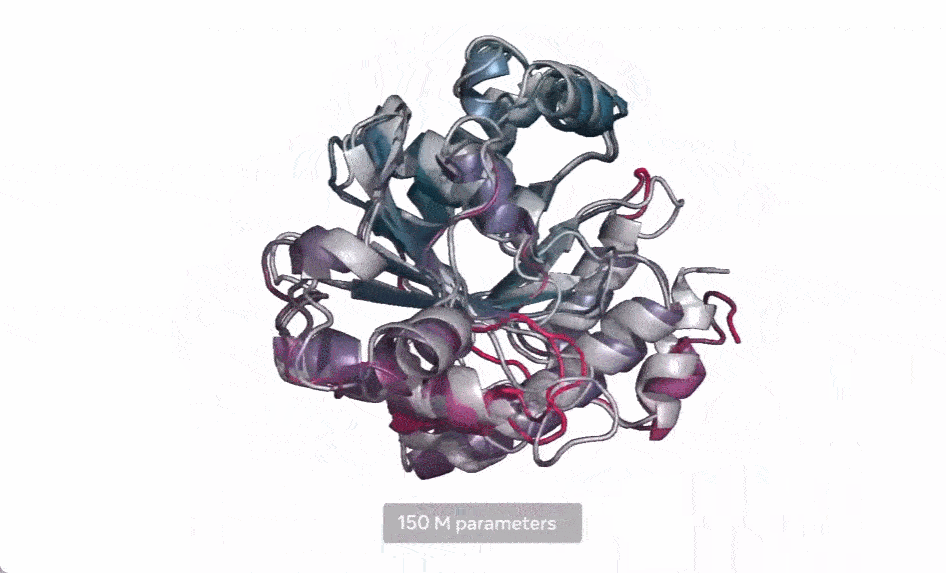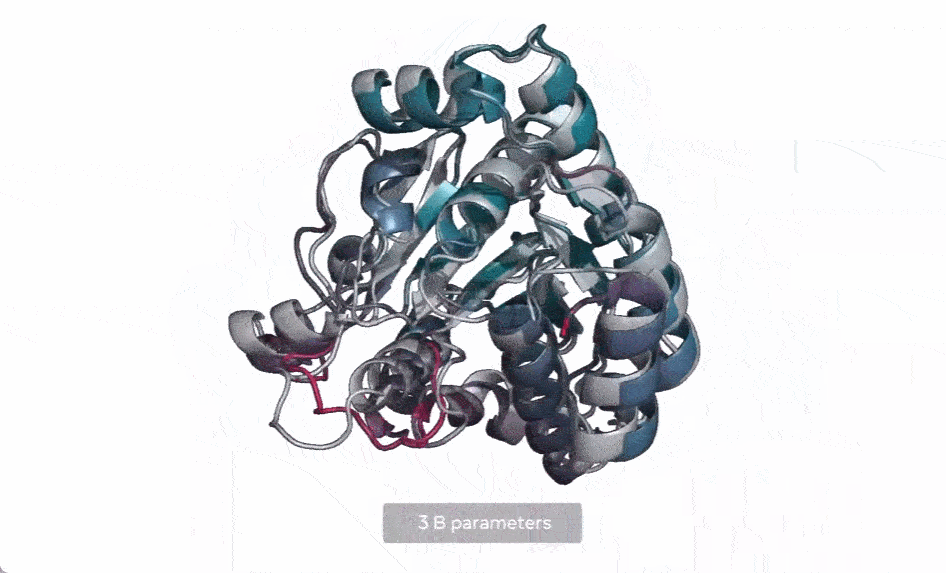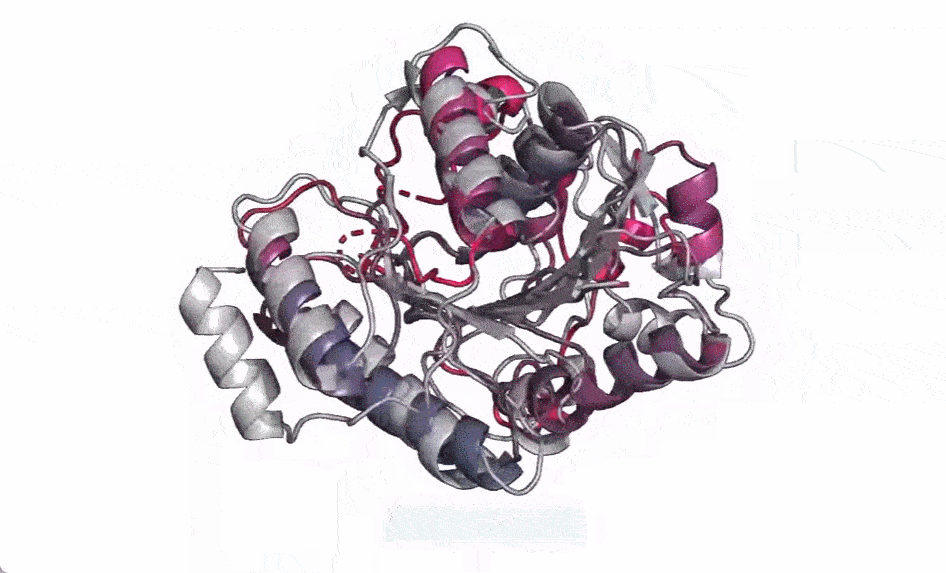
使用當(dāng)前 SOTA 計(jì)算工具,在實(shí)際時(shí)間范圍內(nèi)預(yù)測(cè)數(shù)億蛋白質(zhì)序列結(jié)構(gòu)可能花費(fèi)數(shù)年時(shí)間,即便用上主要研究機(jī)構(gòu)的資源也是如此。因此,想要在宏基因組尺度上進(jìn)行預(yù)測(cè),預(yù)測(cè)速度的突破至關(guān)重要。
Meta AI 發(fā)現(xiàn)使用蛋白質(zhì)序列的語言模型大大加快了結(jié)構(gòu)預(yù)測(cè)的速度,最高提升 60 倍。這足以在短短幾周內(nèi)對(duì)整個(gè)宏基因組數(shù)據(jù)庫做出預(yù)測(cè),并且可以擴(kuò)展到比我們當(dāng)前發(fā)布的數(shù)據(jù)庫大得多的數(shù)據(jù)庫。事實(shí)上,這種新的結(jié)構(gòu)預(yù)測(cè)能力能夠在短短兩周內(nèi),在大約 2000 個(gè) GPU 組成的集群上預(yù)測(cè)超過 6 億多個(gè)宏基因組蛋白的序列。
此外,當(dāng)前 SOTA 結(jié)構(gòu)預(yù)測(cè)方法需要搜索大型蛋白質(zhì)數(shù)據(jù)庫以識(shí)別相關(guān)序列。這些方法實(shí)際上需要一整組進(jìn)化相關(guān)的序列作為輸入,以便它們可以提取與結(jié)構(gòu)相關(guān)的模式。Meta AI 的 ESM-2 語言模型在其對(duì)蛋白質(zhì)序列的訓(xùn)練過程中學(xué)習(xí)這些進(jìn)化模式,進(jìn)而能夠直接從蛋白質(zhì)序列中對(duì) 3D 結(jié)構(gòu)進(jìn)行高分辨率預(yù)測(cè)。
下圖展示了使用 ESM-2 語言模型進(jìn)行蛋白質(zhì)折疊。箭頭從左到右顯示了網(wǎng)絡(luò)中從語言模型到折疊 trunk 再到結(jié)構(gòu)模塊的信息流,最后輸出 3D 坐標(biāo)和置信度。
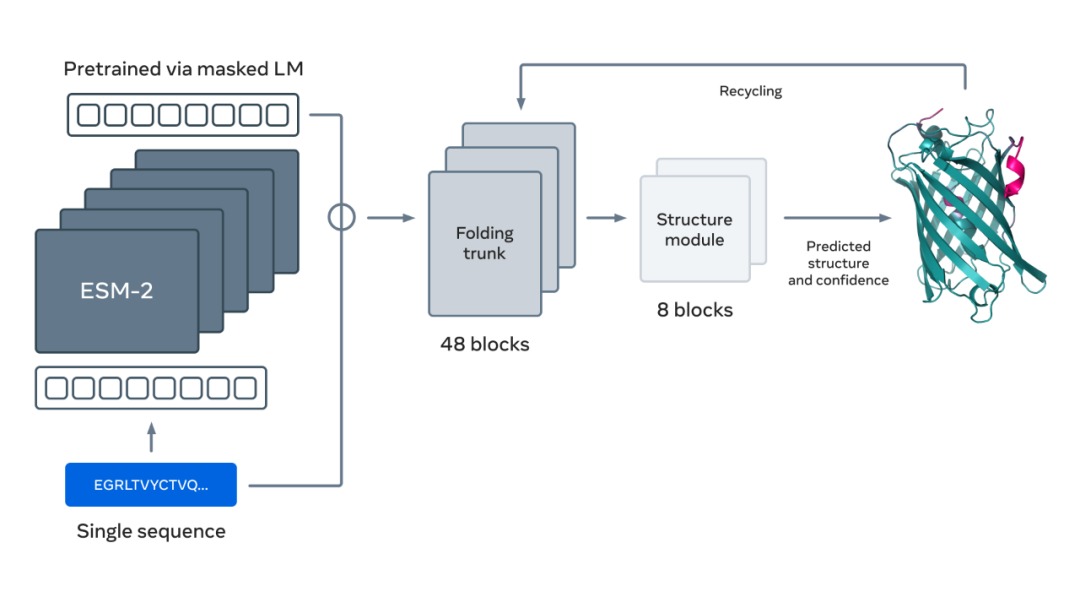
更多詳細(xì)內(nèi)容請(qǐng)參閱原文。
博客鏈接:https://ai.facebook.com/blog/protein-folding-esmfold-metagenomics/